Daten ohne Wissen: die bildgebende Hirnforschung auf dem Prüfstand
Die Neurowissenschaft erlebt nun schon seit Jahrzehnten einen Hype, an den kaum eine andere Disziplin herankommt. Das Gehirn ist das Ornament unserer Zeit, kein Buchcover kommt mehr ohne eines aus. Das liegt nicht zuletzt daran, dass ein essentieller Bestandteil der Hirnforschung auf Bildgebungen beruht, meist mittels funktioneller Magnetresonanztomografie (fMRT). Weil sie durch solche Bilder besser greifbar und darstellbar ist als z.B. die Theoretische Physik, ist die Neurowissenschaft eine ästhetisch ansprechende Disziplin, die sich eben auch zur Vermarktung besonders eignet.
Problematisch wird es dann, wenn die Wissenschaft selbst von diesem Hype erfasst wird. So berichtet eine Studie in Nature, dass die bildgebende Hirnforschung überwiegend mit so winzigen Stichprobengrößen arbeitet, dass ihre gemessenen Effekte null und nichtig sind (1). Aufgepasst: Bildgebende Studien, die Verhalten, Motorik, Sprache, Emotion oder Kognition mit Hirnaktivität assoziieren wollen, benötigen demnach für eine seriöse Analyse 1000 bis mehrere tausend Versuchspersonen. Die durchschnittliche Zahl an Versuchspersonen in Hirnforschung aber ist: 25 (Median). Das zieht tausende Forschungsergebnisse in Zweifel, auf denen wiederum andere Forschung aufbaut – eine wissenschaftliche Katastrophe. Und es stellt sich die Frage: wie konnte es dazu kommen?
Ein Problem aller messenden Wissenschaften
Solche Probleme – Probleme der statistischen Power, also der Generalisierbarkeit auf Basis der Stichprobe – sind nicht auf die Neurowissenschaft begrenzt. Meta-analysen und statistische Modellierungen sehen das größte Problem in der Psychologie, generell aber in den meisten Sozialwissenschaften und teilweise in der Medizin (dort vor allem wenn es um Patientenstudien geht). Dieser berüchtigten Replikationskrise liegt als Hauptfaktor vermutlich ein Power-Defizit der Stichproben zugrunde (2, 3).
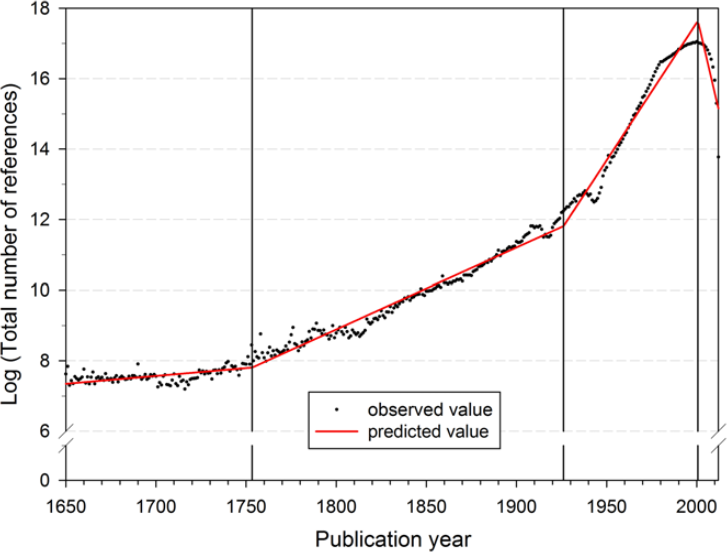
Angesichts der schieren Explosion an Studien, Journals, Arbeitsgruppen und Forschungsmethoden ist es kein Wunder, dass die Qualität der Daten womöglich allmählich ihrer Quantität zum Opfer fällt. Ein Artikel der Max-Planck-Gesellschaft hat den unglaublichen, exponentiellen Zuwachs der Forschungsliteratur eindrucksvoll dokumentiert (Abb. 1).
In einer qualitativen Studie von 2015 wurden Professorinnen und Professoren befragt, wie sich die Forschungslandschaft ihrer Meinung nach verändert habe. Hier ein Auszug aus einem der Interviews:
“Wenn man fünfundzwanzig, dreißig Jahre zurückgeht, als ich in der Ausbildung war, gab es weniger Zeitschriften, die Literatur war kompakter, jetzt gibt es eine größere Anzahl von Zeitschriften und mehr Leute, die um immer weniger Ressourcen konkurrieren, so dass es meiner Meinung nach den Anschein hat, dass eine Menge von dem, was veröffentlicht wird, ziemlich abwegig oder fragwürdig ist“ (5).
Es dürfte allen klar sein: wenn immer mehr Menschen immer mehr publizieren, dann hat man immer weniger Zeit, das Publizierte genau und kritisch zu prüfen. Man generiert mehr und mehr Daten, das Wissen um deren Zuverlässigkeit aber nimmt ab.
Viele Daten um nichts
Kommen wir zum Kern des Problems. Häufig wird behauptet, dass Daten das neue Öl seien. Falls das stimmt, dann gilt aber auch: so wie Öl muss man Daten erst raffinieren, bevor sie nutzbar sind. In der Forschung nennt sich das auch pre-processing. Eine Gefahr, die ich im Forschungsbetrieb selbst beobachten konnte, ist, dass die heutige Allgegenwärtigkeit von Daten suggeriert, man könne spannende Effekte leichter als je zuvor entdecken. Zum Beispiel, indem man Onlineexperimente oder -umfragen durchführt, oder indem man uralte Datensets re-analysiert. Dabei gibt man in solchen Fällen jenes entscheidende Kriterium auf, welches Daten erst ihre Qualität verleiht: die Kontrolle über die Erhebung. Wer weiß denn, wie akkurat die alten Daten damals erhoben worden sind? Wer weiß, wie ernsthaft die Versuchspersonen zuhause das Onlineexperiment bearbeiten?
Aus verzerrten oder fehlerhaften Daten lassen sich keine zuverlässigen Schlüsse ziehen. Doch der Verführung eines leichten Zugangs zu schlechten Daten dürften viele Forscherinnen und Forscher erliegen, schließlich ist eine hohe Publikationsfrequenz in der Wissenschaft zum K.O.-Kriterium der Karriere avanciert.
Fancy, shiny, flashy
In diesem Sinne zeichnet sich, so mein Eindruck, folgendes Bild: auf der Autobahn der innovativen Forschung, wo eine clevere Methode die andere jagt, wo stets eine noch ausgefallenere Technologie dazukommt, muss der Lebenslauf der Wissenschaftlerinnen und Wissenschaftler stets kenntlich machen, dass sie die cutting-edge Methoden ihres Fachs beherrschen, dass sie smart genug sind, dieses oder jenes vielversprechende Instrument zu nutzen. Vor wenigen Jahren war KI noch ein mysteriöser Komplex, heute findet sie sich quasi in jeder neurowissenschaftlichen Masterarbeit (da stellt sich doch die Frage, ob die Menschen in so wenigen Jahren diese Technik gemeistert haben, oder ob KI, wie sie gemeinhin verwendet wird, nicht einfach ein besserer Taschenrechner ist?). Fancy, shiny, flashy scheint das Motto zu sein, wenn es in der Forschung um die Methodenwahl geht.
In einem vielzitierten Artikel in Neuron warnten Krakauer und Kollegen bereits 2017 davor, dass Neurotechnologien nur ein Mittel zum Zweck seien, nicht der Zweck selbst (6). Ohne ein intelligentes Forschungsdesign und gut begründete Hypothesen nützen auch die innovativsten Instrumente nichts. Doch weil gute Methoden und damit Daten heute relativ leicht verfügbar sind, ist es verführerisch, sich darauf zu verlassen und weniger Mühe in die Entwicklung guter Hypothesen zu stecken.
Vorbild Genetik
Zurück zur Ausgangslage. Was kann uns helfen, die bildgebende Hirnforschung wieder auf die Beine zu bringen? Ein Vorbild könnte die Genetik sein, die früher dasselbe Problem hatte. Sogar inhaltlich, insofern als frühe genetische Studien häufig psychiatrische Krankheitsbilder mit einzelnen Genen assoziierten – Korrelationen, die heute meist als abwegig gelten. Stattdessen führen Genetikerinnen und Genetiker heute, auf Basis von Gendatenbanken, Studien mit hunderttausenden bis Millionen Versuchspersonen durch. Auch berücksichtigen sie die Komplexität des menschlichen Genoms in ihren Analysemethoden und Studiendesigns (z.B. Zwillingsstudien).
Diese Fortschritte in der Genetik hatten allerdings auch einen Preis: häufig sind die Autorenlisten der Artikel extrem lang, da für so große Stichproben stets mehrere internationale Teams kooperieren müssen und die Geschwindigkeit der Publikation nimmt natürlich ab. Für verantwortungsbewusste Forscherinnen und Forscher – welche immerhin durch Steuergelder bezahlt werden – sollte die Datenqualität allerdings die erste Priorität sein.
Quellen
(1) Marek, S., Tervo-Clemmens, B., Calabro, F. J., Montez, D. F., Kay, B. P., Hatoum, A. S., … & Dosenbach, N. U. (2022). Reproducible brain-wide association studies require thousands of individuals. Nature, 603(7902), 654-660.
(2) Szucs, D., & Ioannidis, J. P. (2017). Empirical assessment of published effect sizes and power in the recent cognitive neuroscience and psychology literature. PLoS biology, 15(3), e2000797.
(3) Ritchie, S. (2020). Science fictions: Exposing fraud, bias, negligence and hype in science. Random House.
(4) Bornmann, L., & Mutz, R. (2015). Growth rates of modern science: A bibliometric analysis based on the number of publications and cited references. Journal of the association for information science and technology, 66(11), 2215-2222.
(5) Siebert, S., Machesky, L. M., & Insall, R. H. (2015). Overflow in science and its implications for trust. Elife, 4, e10825.
(6) Krakauer, J. W., Ghazanfar, A. A., Gomez-Marin, A., MacIver, M. A., & Poeppel, D. (2017). Neuroscience needs behavior: correcting a reductionist bias. Neuron, 93(3), 480-490.


Interessant :
Die Datenqualität meint insbesondere die Qualität der Erfassung von Daten?
Die Datenqualität kann mit 25 Probanden wie auch mit riiesengroßen erfassten Gendatenhaltungen iO sein, ohne dass sich so Informationsqualität sicher stellen lässt?
MFG – WB
“What I cannot create, I do not understand”
Die funktionelle Bildgebung des Gehirns mittels Kernspintomographie (FMRI) hat die Hirnforschung wunderbar anschaulich gemacht und zu einer Art Behavioristischer Hirnforschung geführt. Man spricht etwa von Neuroökonomie, wenn man den Einsatz von fMRI, Enzephalograhie etc. für die Aufklärung von „ökonomischem Verhalten“ (etwa wie Angst vor Besitzverlust sich im Hirn „ausdrückt“).
Allerdings wird schon lange argumentiert, dass in diesem Gebiet vorschnelle Schlüsse gezogen werden und auch dass die Stichproben viel zu klein sind um zu erhärtbaren Aussagen zu kommen.
Meine Behauptung: Hirnuntersuchungen an Einzelpersonen etwa mit Brain-Computer-Interface werden erst dann zu wissenschaftlich/technisch erhärteten Aussagen kommen, wenn man das, was man misst mit dem was sich anschliessend im Hirn und im Verhalten verändert in Zusammenhang bringt und man zudem sehr grosse Stichproben hat, die es erlauben zwischen individuellen und allgemeingültigen Zusammenhängen zu unterscheiden. Zudem muss der vermutete Zusammenhang dadurch erhärtet werden, dass man nicht nur Hirnaktivität misst, sondern sie auch künstlich (etwa mit Strömen abgegeben von implantierten Elektroden) auslöst.
Begründung: Das Hirn ist äusserst komplex mit sehr vielen Wechselwirkungen zwischen verschiedenen Hirnzentren, wobei sich sogar die Hirnchemie lokal ändern kann. Es sind also dynamische Prozesse mit zeitlicher Entwicklung. Ein einzelnes fMRI-Bild ist deshalb nur ein Schnappschuss in einem ganzen Prozess und genügt nicht um den ganzen Prozess zu verstehen.
Hallo Herr Holzherr,
Sie schreiben u.a.:
“Begründung: Das Hirn ist äusserst komplex mit sehr vielen Wechselwirkungen zwischen verschiedenen Hirnzentren, wobei sich sogar die Hirnchemie lokal ändern kann. Es sind also dynamische Prozesse mit zeitlicher Entwicklung. Ein einzelnes fMRI-Bild ist deshalb nur ein Schnappschuss in einem ganzen Prozess und genügt nicht um den ganzen Prozess zu verstehen.”
Ich weiß nicht ob ich Sie falsch verstehe, aber ich vermute dass Sie nicht verstanden haben was fMRI an Daten überhaupt aufnimmt. Bei MRI sowie bei fMRI wird vereinfacht ausgedrückt der komplette Scanbereich (Kopf, Gehirn, Luft drumherum) in Voxel (3D pixel) eingeteilt.
Für jeden Voxel werden dann über die Zeit hinweg einzelne Datenpunkte aufgenommen, also das BOLD Signal gemessen. Heute geht das mit Aufnahmegeschwindigkeiten von etwa 1 Hz für das gesamte Gehirn bzw. den gesamten Kopfbereich, also 1 Datenpunkt pro Sekunde.
Wenn man nun beispielsweise 900 Sekunden aufnimmt erhält man also 900 Datenpunkte pro Voxel, d.h. man erhält eine time-series (Deutsch: Zeitreihe). Somit erhält man sehr wohl den zeitlichen Verlauf bzw. die Dynamik des BOLD Signals.
Das wovon Sie sprechen, also bunte Aktivierungsmuster auf Gehirnscans, sind sogenannte statistical parametric maps. Diese sind nur eine von quasi unendlich vielen Methoden wie man fMRI Daten auswerten kann. Unter Laien scheint es mir aber häufiger so zu sein dass die Annahme besteht dass diese maps immer das Ergebnis von fMRI Analysen seien, sie werden mit fMRI quasi gleichgestellt. Das ist natürlich ein Fehler.
Zum Thema von David:
Ich glaube nicht dass die Genetik ein Vorbild für die Datenerhebung bzw. Verbesserung der Stichprobengröße (Probandenanzahl) in fMRI sein kann und möchte das kurz und einfach begründen.
Wie das gemessene BOLD Signal sich verhält hängt nicht nur von der Person im Scanner ab die gemessen wird, sondern auch vom Scanner sowie den Scannereinstellungen selbst. Insbesondere die Scaneinstellungen (für die Profis: TR, TE, flip angle, etc.) können das Signal und somit die Rohdaten leicht bis stark beeinflussen. Damit werden auch automatisch viele spätere Messungen der Datenanalyse am Computer mitbeeinflusst.
Was in Scanner A mit Einstellungen Ax gemessen wurde kann also nicht 1:1 mit Ergebnissen verglichen werden die in Scanner B mit Einstellungen Bx herauskamen.
Wenn man also an verschiedenen Standorten für eine Studie Probanden messen möchte (um somit die Stichprobengröße radikal zu vergrößern), dann müsste überall auch mit dem gleichen Scanner, den gleichen Settings, etc. pp gemessen werden. Ansonsten erhält man zwar eine größere Stichprobenzahl, allerdings sind die Daten der einzelnen Personen zwischen den Erhebnungsorten nicht mehr vergleichbar. Dass das in der Praxis schwer umzusetzen ist dürfte klar sein.
Auch können verschiedene Scannersettings jeweils besser oder schlecht für ein bestimmtes finales Analyseziel sein. Man muss also ohnehin Abstriche machen wenn riesige Studien wie das Human Connectome Project durchgeführt werden bei denen die Rohdaten für möglichst viele Analysen passend sein sollen.
Aus diesen und weiteren Gründen ist es deshalb meiner Ansicht nach nicht möglich ein Vorgehen wie in der Genetik für fMRI anzuwenden. Oder sehe ich das zu kritisch?
So ist mE nicht ganz korrekt :
Feynman, nicht wahr, ich habe nicht nachgeschaut.
Besser ist womöglich :
MFG – WB
Gut geschrieben! Allerdings ziehe ich zur Lösung andere Schlüsse aus der Situation. fMRT (Methode meines eigenen PhD) ist, denke ich, gerade darum so erfolgreich, weil sie so schlecht ist. Damit reduziert man Aktivitäten von 87 Milliarden Neuronen im Millisekundentakt auf ein grobes Blutflusssignal von einigen Zehntausend Bildpunkten im Sekundentakt – damit kann man immerhin rechnen.
Wenn man kritischer darüber nachdenkt, was die (Forschungs-) Frage (explanandum) ist, kommt man vielleicht eher auf den Gedanken, die Antwort nicht im Scanner zu suchen. Da Denken und Verhalten immer verköpert und situiert sind, ist die ökologische Validität solcher Experimente a priori niedrig; dafür muss man erst gar nicht über statistische Power nachdenken.
Noch eine Sache zur Anzahl der Publikationen: Die Zitationen, die man auf der Grafik sieht, sprechen für die Zitationsdichte. Das ist kein Beleg für einen Anstieg der Publikationen. Deren Zahl müsste man zudem in Relation zur Zahl der Wissenschaftler*innen setzen (die auch gestiegen ist). Im Ergebnis wird’s aber schon stimmen.
Übrigens kann man auch ein Buch ohne Gehirn auf dem Umschlag schreiben: Auf meinem wächst ein Baum auf einem Kopf.