Wie Gentechnik am Menschen funktioniert
Meine heutige Gastautorin Maike Voges erklärt, wie die neue Technik der Keimbahnmanipulation an menschlichen Embryonen funktioniert und warum das trotzdem alles nicht so einfach ist. Normalerweise forscht sie am Heinrich-Pette-Institut in Hamburg an neuen Therapien gegen Infektionskrankheiten. Momentan arbeitet sie in der AIDS-Forschung und nimmt nebenher an einem Lehrgang für Wissenschaftsjournalismus und Öffentlichkeitsarbeit teil. 
Es ist eine Premiere in der medizinischen Forschung, die Hoffnung macht und gleichzeitig Ängste schürt: zum ersten Mal gelang es Wissenschaftlern in den USA, einen krankheitsauslösenden Genfehler direkt nach Befruchtung menschlicher Eizellen vollständig zu reparieren. Die daraus entstandenen Embryonen sind genetisch nicht mehr von ihren gesunden Geschwisterzellen zu unterscheiden. Dieses spektakuläre Ergebnis veröffentlichte die Forschergruppe um Prof. Dr. Shoukhrat Mitalipov aus dem „Center for Embryonic Cell and Gene Therapy“ der Oregan Health & Science Universität letzte Woche in dem Fachmagazin „Nature“.
In der Studie wurden gesunde Eizellen mit Samen eines Spenders befruchtet, der einen Fehler in einem seiner beiden Gen-Allele MYBPBC3 trägt – eine autosomal-dominante Erbkrankheit. Dieser Gendefekt führt zu einer Fehlentwicklung des Herzmuskels und somit zu einer Herzschwäche (hypertrophe Kardiomyopathie), die häufigste Todesursache junger Sportler.
Die Forscher reparierten diesen Fehler erfolgreich mittels einer noch recht neuen biochemischen Methode namens CRISPR/Cas9 und entdeckten dabei einen völlig unbekannten DNA-Reparaturmechanismus, der offensichtlich nur in Keimbahnzellen – also Samen und Eizellen – vorkommt.
CRISPR/Cas9 schneidet Gene an genau definierten Stellen entzwei
Die CRISPR/Cas9 Technologie funktioniert im Prinzip wie eine zielgerichtete Schere: das Enzym bindet durch eine von den Wissenschaftlern künstlich hergestellte RNA an die gewünschten Stelle im Erbgut und schneidet dort die DNA in zwei Teile. Anschließend baut die Zelle die geschnittene DNA wieder mit Hilfe von einem der beiden internen Reparaturmechanismen zusammen:
- sehr unpräzises Einfügen von kleinen, zufällig ausgewählten DNA-Bestandteilen in den Schnitt („non-homologous end-joining, NHEJ“) oder
- fehlerfreier Einbau eines längeren Strangs ungebundener DNA in das geschnittene Gen („homology-directed repair, HDR“)
DNA Reparaturmechanismen in einer Zelle. Links die unpräzise NHEJ, die häufig zu einer Zerstörung eines Gens führt, rechts die genauere HDR, die zielgenau und spezifisch den Bruch repariert.
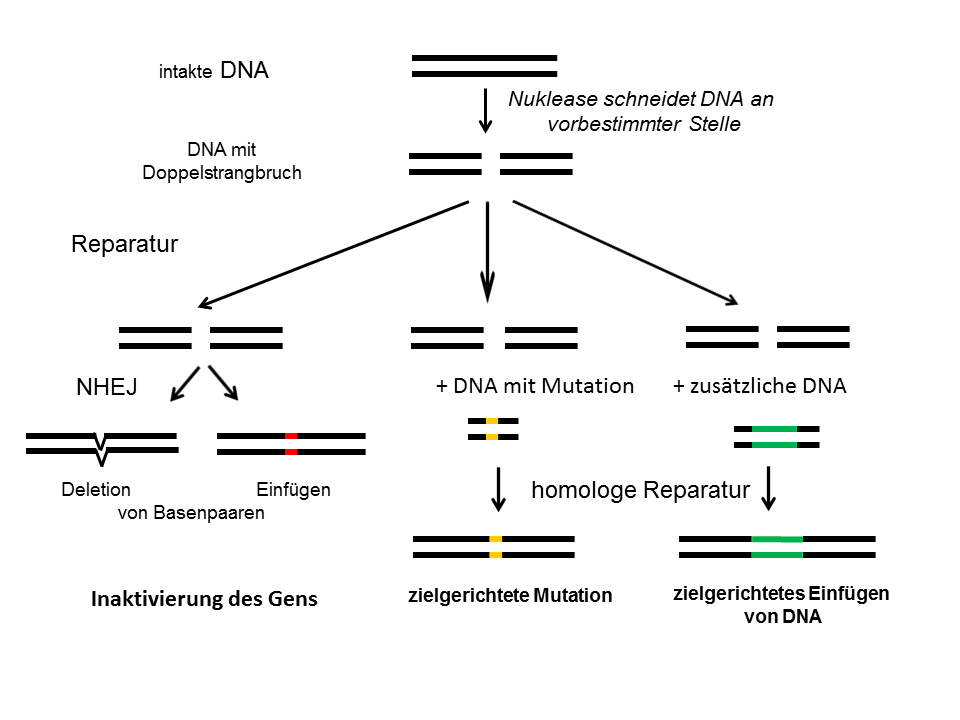
Der unpräzise Mechanismus findet in der Regel (leider) deutlich häufiger statt als der präzise, was eines der großen Probleme der Gentherapie mit CRISPR/Cas9 ist: die ungenaue Reparatur führt zu nicht vorhersagbaren weiteren Mutationen in den Genen. Im besten Fall ist das Gen repariert und danach funktionstüchtig, in den meisten Fällen ist das Gen aber völlig zerstört. Soll also ein Gendefekt repariert werden wie in der vorliegenden Studie, ist der präzise Reparaturmechanismus unerlässlich, während der unpräzise Mechanismus möglichst nicht stattfinden darf.
CRISPR/Cas9 repariert den Gendefekt in Keimbahnzellen präziser als erwartet
Und genau das war auch die große Überraschung in den Ergebnissen der Studie: in Keimbahnzellen wird die geschnittene DNA in den allermeisten Fällen höchst präzise wieder zusammengebaut.
In den ersten Versuchen schleusten die Wissenschaftler die Gen-Schere CRISPR/Cas9 zusammen mit einer künstlich hergestellten Ersatz-DNA in isolierte Stammzellen des kranken Spenders ein. Die Ersatz-DNA enthält die korrekte Gensequenz des MYBPBC3 Gens und wird im Idealfall an der Schnittstelle eingebaut („Knock-In“), um den Gendefekt zu beheben. Bei der Analyse zeigte sich, dass nur in 41% der überhaupt reparierten Zellen die präzise Korrektur des Gendefekts durch Einbau der Ersatz-DNA stattfand, in den anderen Zellen wurde die geschnittene DNA willkürlich und unpräzise wieder zusammengefügt.
Ähnliches erwarteten sie nun auch bei ihren nächsten Versuchen in Keimbahnzellen. Sie befruchteten gesunde Eizellen mit dem Samen des kranken Samenspenders und schleusten die Gen-Schere zusammen mit der künstlichen DNA ein. Die befruchteten Eizellen entwickelten sich dann über die nächsten Tage im Brutschrank zu Blastozysten (eine embryonale Vorstufe), die anschließend von den Forschern analysiert wurden.
Zu ihrer großen Überraschung fanden die Forscher in den Embryonen viel mehr präzise korrigiertes Erbgut (72%), während die unpräzise Reparatur deutlich reduziert vorkam (28%)! Ebenfalls spektakulär ist, dass die präzisen Reparaturmechanismen nicht (wie sonst üblich) die vorhandene Ersatz-DNA in das geschnittene Gen einbauten. Stattdessen diente das andere gesunde Allel der Mutter als Vorlage für eine DNA-Neusynthese in der Zelle: die Keimbahnzellen haben offensichtlich einen eigenen, viel genaueren Reparaturmechanismus. Auch ein weiteres großes Problem der Gen-Editierung mit der CRISPR/Cas9 Methode ist von den Forschern in dieser Studie ausgeschlossen worden: die sogenannten „Off-targets“ also das Zerschneiden von ähnlichen DNA-Sequenzen an anderen Stellen im Erbgut. Die Wissenschaftler isolierten die gesamte genomische DNA aus den behandelten Blastozysten und überprüften diese auf falsch gesetzte Schnitte an potentiellen „Gefahrstellen“, fanden aber keine „Off-Targets“. Das Enzym hat also nur die gewünschte Stelle im Erbgut zerschnitten.
Der Startzeitpunkt der Therapie ist entscheidend
Zusätzlich entdeckten die Forscher in dieser Studie, dass der Zeitpunkt der Zugabe der Gen-Schere entscheidend ist. Vorherige Studien in China scheiterten unter anderem an Mosaik-Blastozysten, also an unterschiedlichem Erbgut in den verschiedenen Zellen eines Embryos. Auch die US-Forscher erhielten in 54 herangereiften Embryonen 8 Mosaike, wenn sie zuerst die Eizelle befruchteten und wenig später die Gen-Schere samt künstlicher DNA in die Eizelle einschleusten.
Als Ursache vermuteten sie, dass das Erbgut sich bis zum Eintreffen der Gen-Schere verdoppelt hatte: somit lagen in der Zelle schon zwei defekte Allele vor, die dann unabhängig voneinander repariert wurden (oder auch nicht). Diese beiden nun voneinander unterschiedlichen Allele teilten sich dann im nächsten Schritt auf zwei Zellen auf, die ursprüngliche Zelle unterschied sich also genetisch von der Tochterzelle. Beide Zellen teilten sich weiter und die Mosaik-Embryonen entstanden.
Um dies im nächsten Versuch zu vermeiden, injizierten die Wissenschaftler die Gen-Schere und die künstliche DNA gleichzeitig mit dem Samen in die Eizelle. Das Erbgut lag zu diesem Zeitpunkt auf keinen Fall doppelt vor, die Gen-Schere reparierte umgehend das eine defekte Allel und es entstand von 42 herangereiften Embryonen nur ein einziges Mosaik, eine deutliche Verringerung im Vergleich zu dem vorherigen Versuch. Somit ist klar: der Zeitpunkt ist entscheidend, je früher desto besser!
Medizinisch nur selten relevant und ethisch bedenklich
Die erfolgreichen Ergebnisse dieser Studie erwecken Hoffnung auf eine Zukunft ohne Erbkrankheiten. Was auf den ersten Blick aber wie ein großer medizinischer Durchbruch scheint, ist eigentlich keiner. In unseren Zellen liegt unser Erbgut immer doppelt vor, jedes Gen ist zweifach vorhanden. Die Keimzellen bilden eine Ausnahme, in ihnen liegt jeweils nur eines der beiden Chromosomen-Allele vor, da bei der Befruchtung der Eizelle von beiden Partnern Erbgut an die Nachkommen weitergeben wird. Somit gibt der Gendefekt-Träger bei Erbkrankheiten, in denen nur ein Chromosom betroffen sein muss für die Symptome (autosomal-dominaten Erbkrankheiten) das „kranke“ Gen mit einer Wahrscheinlichkeit von 50% an seine Nachkommen weiter.
Somit ist die Hälfte der erzeugten Embryos sowieso gesund, während die andere Hälfte das defekte Gen enthält. Durch die Behandlung mit CRISPR/Cas9 (nach künstlicher Befruchtung) erhöhte sich der Anteil der gesunden Embryonen von 50% auf 70%. Medizinisch hat das jedoch kaum Bedeutung: bei Vorliegen einer schweren Erbkrankheit in der Familie ist es möglich, nach künstlicher Befruchtung die Embryonen auf mögliche Defekte zu testen und nur die gesunden Embryonen in die Gebärmutter zu implantieren.
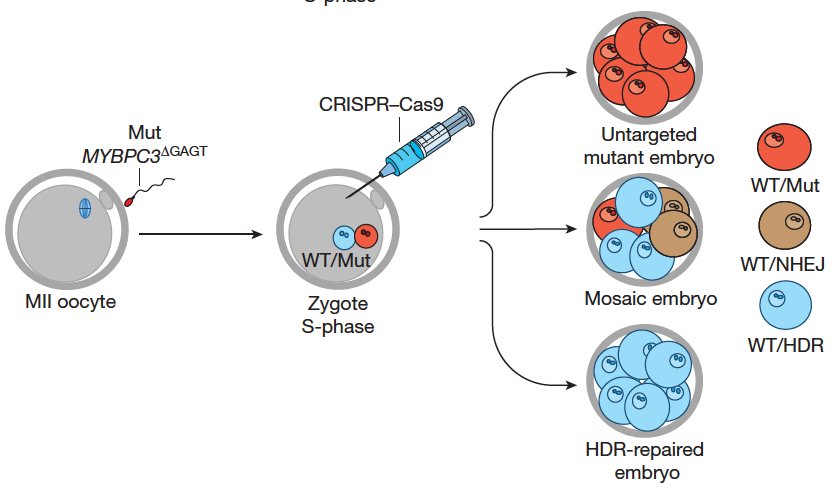
Quelle: Hong Ma, Nuria Marti-Gutierrez, Sang-Wook Park, Jun Wu et al.,
Correction of a pathogenic genemutation in human embryos,, Advance Online Publication, 2017, DOI: 10.1038/nature23305, Fig. 1
So richtig interessant wird die Technologie für die Therapie von autosomal-rezessiven Erbkrankheiten. Nur wenn der Gendefekt auf den beiden identischen Chromosomen vorliegt, entwickelt sich eine Krankheit. Ein bekanntes Beispiel dafür ist die Stoffwechselerkrankung Mukoviszidose, die häufigste tödlich verlaufende genetische Erkrankung in der hellhäutigen Bevölkerung. Hier vererben Erkrankte den Gendefekt grundsätzlich immer an die nächste Generation weiter, denn keines der beiden vorliegenden Gene ist intakt. Für die Betroffenen ist eine große psychische Belastung und viele entscheiden sich trotz Kinderwunsch gegen eigene Kinder. CRISPR/Cas9 könnte hier gezielt das kranke Gen in der befruchteten Keimbahnzelle mit Hilfe des gesunden Genes des Partners reparieren und somit eine Übertragung der Erbkrankheit an die Folgegenerationen verhindern.
Aber die genetische Veränderung des Erbguts in Keimbahnzellen – also Eizellen und Samenzellen – ist zu recht höchst umstritten und ethisch bedenklich: die hier künstlich eingefügten Korrekturen werden an alle weiteren Folge-Generationen vererbt, die Risiken nicht abzusehen.
Viel weitere Forschungsarbeit nötig und trotzdem keine Designerbabys in Aussicht
Die hier entdeckte viel präzisere Korrektur der DNA-Schäden in Keimbahnzellen ist eine sehr gute Nachricht für die CRISPR/Cas9 Methode, da der normalerweise häufiger auftretende unpräzise Reparaturmechanismus eines der großen Probleme in der potentiellen medizinischen Anwendung ist. Die Entwicklung der Technologie für medizinische Zwecke ist somit einen großen Schritt weiter gekommen! Die sehr junge CRISPR/Cas9 – Technologie benötigt aber trotzdem noch einiges an Forschungsarbeit, um detailliert zu verstehen, was eigentlich genau in den Zellen passiert und wie die unerwünschten Nebeneffekte verhindert werden können. In den letzten Jahren arbeiteten einige Labore weltweit daran, dass Cas9 Protein weiterzuentwickeln. Eine der Weiterentwicklungen, die Cas9-Nickase, bindet sehr viel genauer an das Erbgut verringert somit die „Off-target“ Problematik, zusätzlich repariert die Zelle die von der Nickase gesetzten Schnitte häufiger mit dem präzisen Mechanismus als bei Schnitten, die mit dem ursprünglichen Enzym entstanden.
Es wird interessant zu sehen, wie gut die neue Cas9 Nickase in Keimzellen arbeitet und ob es möglich ist, damit auch dominant-rezessive Krankheiten zu heilen. Für die Gegner der Technologie gibt es ebenfalls eine gute Nachricht: nur was genetisch auf dem anderen vorliegenden Chromosom festgelegt ist, kann für eine Reparatur des „kranken“ Gens genutzt werden. Die Entwicklung der gefürchteten „Designerbabys“ ist also ein ganzes Stück schwerer als gedacht und die „Schöne Neuen Welt“ von Huxley zumindest vorerst noch in weiter Ferne – dennoch sind die Risiken nicht abzusehen.


Könntest du vielleicht diese Aussage noch genauer erklären: “Stattdessen diente das andere gesunde Allel der Mutter als Vorlage für eine DNA-Neusynthese in der Zelle: die Keimbahnzellen haben offensichtlich einen eigenen, viel genaueren Reparaturmechanismus. ” Gibt es zu diesem Mechanismus schon Vermutungen, wie er funktioniert?
Was bedeutet es außerdem, wenn eine Gen “zerstört” wird wie in der NHEJ?
Hallo Mathilde,
das läuft über homologe Rekombination, heißt also die beiden Allele werden nahe zusammengebracht und dann das kaputte Stück anhand des gesunden Allels repariert: https://de.m.wikipedia.org/wiki/Rekombination_(Genetik)
Richtig neu ist, dass hier lieber dieser Mechanismus angeschaltet wird statt die mit eingefügte freie synthetische DNA einzubauen. Das ist in anderen Zellen ganz anders.
Und zu deiner zweiten Frage: durch die willkürlich eingefügten DNA Bestandteile kommt es zu Mutationen im Gen, zum Beispiel ein Frame Shift oder es wird ein Stopp eingefügt. Das dann gebildete Protein ist dann entweder kürzer weil es an der “zerstörten” Stelle abbricht oder es kommt zu einem ganz anderen Protein mit veränderter Struktur, andere Aminosäuren werden eingebaut. Ich hoffe, die Antwort hilft weiter 🙂
Viele Grüße,
Maike
Hallo Scilogs!
Schöner Artikel, jedoch haben sich ein paar Ungenauigkeiten und Fehler eingeschlichen.
Einen guten Überblick (samt Schemata) zur Funktionsweise (und auch zu den experimentellen Grundlagen) von CRISPR/Cas9 findet man auf der Internetseite von “addgene” (1). Darüber hinaus habe ich bereits zum Spiegelartikel (2), der sich mit der Nature-Publikation befasste, meinen Senf dazugegeben (3).
Nun aber zurück zu Ihrem Artikel:
Scilogs: “1. sehr unpräzises Einfügen von kleinen, zufällig ausgewählten DNA-Bestandteilen in den Schnitt („non-homologous end-joining, NHEJ“) oder”
>> Bei der Reparatur eines Doppelstrangbruches (DSB) durch NHEJ geschieht dies meist ohne Schaden (4) (5) und in “nur” etwa 5% der Fälle (4) werden kurze “INDELs” (insertion or deletion) erzeugt. Beim “genome editing” werden jedoch akkurat reparierte Abschnitte (im Gegensatz zu INDEL-Abschnitten) wiederholt geschnitten und repariert, wodurch sich mit der Zeit Zellen mit INDEL-Abschnitten anreichern (4). Darüber hinaus agieren bei der Reparatur mitunter auch Exonukleasen, die TdT (“terminal deoxyribonucleotide transferase”) und Pol-µ, die INDELs hervorrufen (5).
Scilogs: “2. fehlerfreier Einbau eines längeren Strangs ungebundener DNA in das geschnittene Gen („homology-directed repair, HDR“)”
>> Das stimmt nur zum Teil. Die homologe DNA (ob nun vom intakten Allel oder vom exogenen Reparaturkonstrukt) dient als Vorlage und wird dann bei der Komplexauflösung teilweise “verbaut”. Bei der Reparatur eines DSB durch HDR werden u.a. die DNA-Enden prozessiert und DNA-Überhänge generiert, die ihrerseits mit komplementärer DNA als Vorlage für eine DNA-Polymerase dienen, um die entstandene Lücke zu füllen (6)(7). Die genauen Mechanismen zum Auflösen der dabei entstehenden Strukturen werden noch untersucht (8).
Scilogs: “…, in den anderen Zellen wurde die geschnittene DNA willkürlich und unpräzise wieder zusammengefügt.”
>> Trotz des Vorhandenseins von INDELs erfolgte das Zusammenfügen keineswegs “willkürlich”, sonst würden “Translokationen” (9) auftreten.
Scilogs: “Zu ihrer großen Überraschung fanden die Forscher in den Embryonen viel mehr präzise korrigiertes Erbgut (72%), während die unpräzise Reparatur deutlich reduziert vorkam (28%)!”
>> Die aus, wie ich vermute, Abb. 2e (10) entnommenen Zahlen beziehen sich auf die “targeting efficiency”, also darauf, welcher Prozentsatz erfolgreich geschnitten und dann mittels HDR oder NHEJ repariert wurde. Von den 72% (13/18) erfolgreich “getroffenen” Embryonen (Mosaik) wurden 55 Blastomere entnommen von denen 63,6% (35/55) durch HDR mittels maternalem (Wildtyp) Allel repariert wurden.
Das Problem dabei, wie die Autoren später selber schreiben, ist, dass die HDR-Berechnung lediglich auf den Mosaik-Embryos beruht, da uniforme WT/WT-Embryos, die auf eine erfolgreiche Reparatur mit dem maternalen Allel zurückgehen, nicht von den homozygoten WT-Embryos zu unterscheiden sind. Tatsächlich ist ein entsprechender Zuwachs von 47,4% dieses WT/WT-Genotyps in unbehandelten zu 66,7% in behandelten Embryos zu verzeichnen (Abb.2a & 2f (10)).
Scilogs: “Auch ein weiteres großes Problem der Gen-Editierung mit der CRISPR/Cas9 Methode ist von den Forschern in dieser Studie ausgeschlossen worden: die sogenannten „Off-targets“ also das Zerschneiden von ähnlichen DNA-Sequenzen an anderen Stellen im Erbgut.”
>> Sie konnten “Off-targets” FÜR diese Studie (weitestgehend) ausschließen. In anderen Studien (mit anderen Eingriffspunkten) stellen diese nach wie vor ein Problem dar (11). Jedoch gibt es auch allgemein Fortschritte bei der Steigerung der Spezifität (12).
Scilogs: “Aber die genetische Veränderung des Erbguts in Keimbahnzellen – also Eizellen und Samenzellen – ist zu recht höchst umstritten und ethisch bedenklich: die hier künstlich eingefügten Korrekturen werden an alle weiteren Folge-Generationen vererbt, die Risiken nicht abzusehen.”
>> Meine Meinung: Was für die “künstlich eingefügten Korrekturen” gilt, gilt auch für spontan auftretende Veränderungen/Mutationen (sofern die Methode nicht großflächig eingesetzt wird, versteht sich). Entweder führen diese zu nicht/vermindert lebensfähigen Nachkommen, zu erstrebenswerten oder zu unerwünschten Eigenschaften (Stichwort: Erbkrankheiten), die entweder durch Verhütung & Adoption oder durch Präimplantationsdiagnostik vermieden oder schlicht als mögliches Risiko in Kauf genommen werden.
Scilogs: “Eine der Weiterentwicklungen, die Cas9-Nickase, bindet sehr viel genauer an das Erbgut verringert somit die „Off-target“ Problematik, …”
>> Die Cas9-Nickase bindet mitnichten “genauer” an das Erbgut. Stattdessen schneidet sie, im Gegensatz zur Standard-Cas9, wie der Name impliziert, nur einen Strang der doppelsträngigen DNA und erzeugt dadurch einen “nick”. Damit daraus ein Doppelstrangbruch wird, bedarf es eines “nick” auf beiden DNA-Strängen möglichst nah beieinander. Zwei “nicks” wiederum bedeutet zwei verschiedene gRNAs (guideRNA) und dies bedingt die höhere Spezifität (13).
Scilogs: “… nur was genetisch auf dem anderen vorliegenden Chromosom festgelegt ist, kann für eine Reparatur des „kranken“ Gens genutzt werden.”
>> Falsch, da man, wie auch im hier besprochenen Nature-Paper (10) angedeutet, Fremd-DNA mit Homologie-Armen/-Flanken (z.B.: “exogenous single-stranded oligodeoxynucleotide (ssODN) templates encoding homology arms to the targeted region” (10)) verwenden kann (14).
MfG, Egooist
(1) http://www.addgene.org/crispr/guide/
(2) http://www.spiegel.de/wissenschaft/mensch/computer-und-dna-die-erbgut-hacker-a-1163546.html
(3) http://www.spiegel.de/forum/wissenschaft/computer-und-dna-die-erbgut-hacker-thread-641418-9.html#postbit_57862141 (Kommentare #87 & #88)
(4) http://blog.addgene.org/crispr-101-non-homologous-end-joining
(5) https://biology.stackexchange.com/questions/38828/how-does-nhej-cause-indels
(6) https://en.wikipedia.org/wiki/Homology_directed_repair#Biological_pathway
(7) https://www.jci.org/articles/view/72992/figure/1
(8) http://www.g3journal.org/content/7/4/1191 (Figure 1)
(9) https://de.wikipedia.org/wiki/Translokation_(Genetik)
(10) http://www.nature.com/nature/journal/v548/n7668/full/nature23305.html
(11) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4338555/pdf/nihms657677.pdf
(12) https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4402539/pdf/gkv226.pdf
(13) https://www.addgene.org/crispr/zhang/faq/
(14) http://www.spiegel.de/video/gentechnik-maeuse-mit-gruenen-fuessen-video-1807613.html
Hallo Scilogs!
Hier noch ein kleiner Nachtrag/Korrektur/Präzisierung zum letzten Punkt meines vorangegangenen Kommentars:
Scilogs: “… nur was genetisch auf dem anderen vorliegenden Chromosom festgelegt ist, kann für eine Reparatur des „kranken“ Gens genutzt werden.”
>> Falsch, da man, wie auch im hier besprochenen Nature-Paper (10) angedeutet, Fremd-DNA mit Homologie-Armen/-Flanken verwenden kann. Wobei das timing (Metaphase II), der Zelltyp (menschliche Oozyte/Zygote) und/oder die Form des eingebrachten Reparaturkonstrukts (exogenous single-stranded oligodeoxynucleotide (ssODN)) in dieser Arbeit eindeutig die HDR durch das mütterliche Allel begünstigen (10). Auch wenn einige Wissenschaftler (z.B. Robin Lovell-Badge) daher meinen, dass “concerns about designer babies – … – may be eased somewhat” (15), zeigen zahlreiche andere Publikationen (16) und u.a. der kurze Beitrag auf SPON (14), dass das “genome editing” von Organismen mit Fremd-DNA prinzipiell gut funktionieren kann. Auch wenn die Effizienz häufig noch zu wünschen übrig lässt.
MfG, Egooist
(10) http://www.nature.com/nature/journal/v548/n7668/full/nature23305.html
(14) http://www.spiegel.de/video/gentechnik-maeuse-mit-gruenen-fuessen-video-1807613.html
(15) https://www.nature.com/news/crispr-fixes-disease-gene-in-viable-human-embryos-1.22382
(16) https://www.ncbi.nlm.nih.gov/pubmed/?term=genome+editing+animals