Künstliche logische Systeme mit Nukleinsäuren
BLOG: Bierologie


 Philipp sagt:
Philipp sagt:
Heut gehts mal um klassische Aussagenlogik, wie sie ein paar Leute hier (inklusive mir selbst) noch in der Oberstufe gelernt haben, angewandt auf lebende Systeme.
Im Kern gehts um das künstliche Erzeugen von logischen Systemen im Lebendigen, also Einrichtungen, die Daten aufnehmen und sie mithilfe logischer Verknüpfungen wie „UND“, „ODER“ etc. verarbeiten (mehr dazu bei Wikipedia).
Hier noch schnell eine kurze Zusammenfassung, z.B. läuft ein logisches UND so ab: Ist der Himmel grau UND die Luft so merkwürdig nass, liegt „Regen“ vor. Anders geschrieben : Grauer Himmel UND Nass = Regen.
Ist der Himmel dagegen grau und die Luft trocken, so liegt natürlich kein Regen vor, es müssen also beide Zustände „wahr“, d.h. in diesem Fall vorhanden sein, damit Regen vorliegt.
Bei einem ODER dagegen muss nur einer der beiden Zustände „wahr“ sein – das sähe beispielsweise so aus: Grauer Himmel ODER Kalt = Schlechtes Wetter; in unserem pessimistischen Beispiel muss der Himmel also nur grau sein, damit das Wetter schon als schlecht gilt.
Dass solche Systeme in vitro (also im Reagenzglas) funktionieren, war schon seit 2004 bekannt. „Damals“ wurde ein DNA-basiertes System benutzt, dass allerdings noch nur über einfache Aussagen funktionierte, es konnte also nur sagen ob die Konzentration eines bestimmten Stoffes hoch oder niedrig (= 1 oder 0) sein konnte. Als Beispiel wurden Indikatoren für Prostatakrebs genommen; damit „der Computer“ angeht muss die BotenRNA der Proteine PPAP2B und GSTP1 in niedriger und PIM1 und HPN in hoher Konzentration vorliegen. Den Computer an sich kann man sich wie eine längere Haarnadel aus DNA vorstellen, an deren Kopf die noch inaktive Droge sitzt.
Die „Arme“ der Haarnadel hängen aufgrund von komplementären Basenpaarungen aneinander; kleine Stückchen der Haarnadel werden dann, je nachdem ob das Level der mRNA hoch oder niedrig ist, Schritt für Schritt abgeschnitten und auf konkurrierende mRNA-Stückchen übertragen.
So wird die Haarnadel Schritt für Schritt kleiner, bis nur noch der Kopf übrig bleibt. Dieser ist allein gesehen nur ein Stück einzelsträngiger DNA, die dann (mehr oder weniger) aufgrund der Komplementärbasenpaarung an die BotenRNA des MDM2-Proteins bindet und so verhindert, dass es als Vorlage für das passende Protein genutzt werden kann.
MDM2 ist in der Zelle verantwortlich für die negative Regulation (also das Runterschrauben) des Tumorrepressorproteins p53, mit MDM2 in der Zelle entwickelt sich also ein Tumor.
Leider ist das ganze nur ein „proof-of-concept“; ob es in der lebenden Zelle funktioniert bleibt noch offen.
2006 wurde dann in Science (Link hier) ein System vorgestellt, bei dem Ein- und Ausgabe in gleicher Form sind – dies ist vor allem für verschieden starke Signale wichtig, da so der Kreislauf mehrmals hintereinander durchlaufen werden kann.
Das System gibt entweder Fluoreszenz ab oder neue Moleküle aus, welche dann wiederum auf andere Systeme Einfluss ausüben können. Wie es im Detail abläuft erkläre ich anhand der nächsten Graphik*:
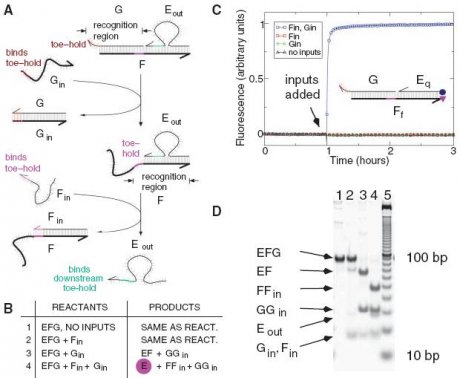
In diesem Bild ist die einfachste Art einer UND-Verknüpfung dargestellt. Unter A sehen wir die logische Schaltung: Ein Molekül, das aus den drei DNA-Strängen G, Eout und F besteht, die aufgrund von Basenpaarung relativ stabil aneinander hängen. Die logische Schaltung lautet: Gin UND Fin = Eout wird freigesetzt.
Wie man sieht hängt von G ein kleines ungepaartes Stückchen namens toe-hold (grob übersetzt Ansatzpunkt) ab – kommt jetzt Gin dazu (also der erste Part unserer Schaltung) so bindet G and Gin, da diese Bindung wesentlich stabiler ist und in der Natur stets nach dem energieärmsten (=stabilsten) Zustand strebt. Diesen G/Gin-Doppelstrang kann man dann vernachlässigen, wir können ihn als Müll betrachten.
Ebenso läufts wenn Fin dazukommt: Es entsteht wieder ein F/Fin-Doppelstrang, so dass nur noch Eout alleine übrig bleibt. B zeigt, welche Zutaten welchen Kuchen ergeben.
Bei C sieht man die experimentelle Bestätigung des Mechanismus durch Fluoreszenz; hier wurden F und Eout mit Fluoreszenzpartikeln versehen, so dass sie beim Freiwerden Licht abgeben, was sich aus der Graphik leicht ablesen lässt. Nur wenn Fin und Gin vorliegen kommt es zu Fluoreszenz.
Abbildung D zeigt eine Gelelektrophorese als Zweitbeweis für die Wirksamkeit des Systems. Die Wissenschaftler haben in ihrer Veröffentlichung noch kompliziertere Systeme ausprobiert und hinbekommen – die noch zu beschreiben sprengt aber den Rahmen, da ich noch ein drittes System vorstellen wollte, welches in meinen Augen das schönste ist – und es hat sogar in Hefezellen funktioniert.
Es wurde in der neuesten Science-Ausgabe unter dem Titel „Higher-Order Cellular Information Processing with Synthetic RNA Devices“ von Maung Nyan Win und Christina D. Smolke vorgestellt.
Dieses System besteht, wie der Name schon sagt, aus 3 künstlichen RNA-Komponenten, einem Aptamer (also einer kurzen RNA-Sequenz, welche spezifisch an ein bestimmtes Signal-Molekül binden kann), einem Ribozym (ein RNA-Molekül, welches wie ein Enzym funktioniert und sich in diesem Falle selbst zerschneidet) und einer Transmitterkomponente, welche alle Teile verbindet.
Weiterhin ist das Ribozym an ein bestimmtes Gen gekoppelt.
Bindet nun ein Signalmolekül an das Aptamer, so verändert der gesamte Kasten seine Konformation und wird so „aktiviert“, ähnlich wie ich meine Schulter bewege, weil ich mit der Hand etwas greife.
Je nachdem wie man es dann haben möchte schneidet das Ribozym sich selbst mit dem Gen, an welches es gebunden wurde, oder es schneidet eben nicht und das Gen wird aktiviert.
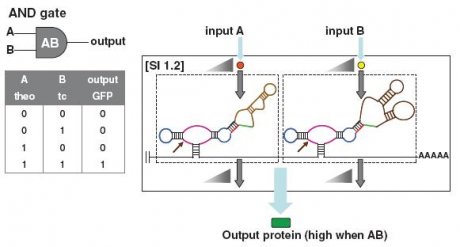
Das nächste Bild** zeigt wieder eine einfache UND-Schaltung.
Die braune Region ist die Aptamerregion an die das Molekül bindet, die blaue und rosa Region ist die Ribozymregion. Nur wenn beide Signalmoleküle an die Vorrichtungen binden kann das Gen aktiviert werden und ein Ausgabeprotein entsteht. Natürlich lassen sich auch zwei oder mehrere Aptamere an ein Ribozym binden, so dass eine simplere UND-Schaltung entsteht.
Die Implikationen von allen drei Techniken sind weitreichend – nicht nur beim Einsatz in „intelligenten“ Medikamenten, wie es schon beim ersten Beispiel erörtert wurde, sondern auch z.B. bei der wirksamen Kontrolle von Mikroorganismen, vielleicht könnte man mit diesem System Mikroorganismen vor Selbstvergiftung schützen, oder künstliche Stoffwechselwege auf andere Teilschritte umschalten lassen sobald ein bestimmtes Signalmolekül vorhanden ist. Da es sich hier um die einfachsten logischen Verknüpfungen handelt kann ich mir auch größere, kompliziertere Biocomputer vorstellen – das ist allerdings noch Zukunftsmusik.
* Diese Graphik wurde von Georg Seelig, et al. erstellt und erschien zusammen mit ihrem Paper „Enzyme-Free Nucleic Acid Logic Circuits“ in Science 314, Seite 1586 (2006)
** Diese Graphik wurde von Maung Nyan Win und Christina D. Smolke erstellt und erschien zusammen mit dem Paper „Higher-Order Cellular Information Processing with Synthetic RNA Devices“ in Science 322, Seite 458 (2008)
Yaakov Benenson, Binyamin Gil, Uri Ben-Dor, Rivka Adar, Ehud Shapiro (2004). An autonomous molecular computer for logical control of gene expression Nature, 429 (6990), 423-429 DOI: 10.1038/nature02551
G. Seelig, D. Soloveichik, D. Y. Zhang, E. Winfree (2006). Enzyme-Free Nucleic Acid Logic Circuits Science, 314 (5805), 1585-1588 DOI: 10.1126/science.1132493
M. N. Win, C. D. Smolke (2008). Higher-Order Cellular Information Processing with Synthetic RNA Devices Science, 322 (5900), 456-460 DOI: 10.1126/science.1160311