Was Ziegenmilch mit genome editing zu tun hat
BLOG: Von Menschen und Mäusen

Jeden Morgen wenn ich zur Arbeit fahre, komme ich an zwei Ziegen vorbei, die hier auf dem Gelände gehalten werden. Ich vermute, dass sie zur Antikörperproduktion gebraucht werden. Ich freue mich jedenfalls immer wenn ich die beiden sehe. Ziegen sind lustig. Hier Beweisstück A:

Ziegen sind aber nicht nur lustig, sondern auch durchaus nützlich. Z.B. habe ich schon gehört, dass man versucht Ziegenmilch als Ersatz für Kuhmilch zu verwenden, um eventuelle Unverträglichkeiten zu vermeiden, obwohl Ziegenmilch natürlich auch Lactose enthält.
In dieser Nature-Veröffentlichung einer chinesischen Gruppe ging es darum, Ziegen genetisch so zu verändern, dass sie das β-Lactoglobulin (BLG) nicht mehr bilden. Dieses Milchallergen kommt auch in Kuhmilch vor und spielt eine Rolle bei der Entwicklung einer Milchallergie (nicht zu verwechseln mit einer Lactoseintoleranz, obwohl die Symptome ähnlich sind). Ziel dieser Arbeit war nicht unbedingt die Produktion BLG-freier Milch zum Verzehr. Laut eigener Aussage geht es der Gruppe dabei eher um eine weitere Untersuchung der Allergenität von BLG und darum seine biologische Funktion zu klären. Darüber hinaus wurde an der Stelle, an der vorher die genetische Sequenz für das BLG war, die Sequenz des humanen Lactoferrin (ein Protein, dass beim Eisentransport im Intestinaltrakt wichtig ist) eingebracht.
Ein Nachbar hier auf Scilogs hat mir empfohlen über mein eigenes Forschungsgebiet zu schreiben, also möchte ich diesen Rat zumindest für den heutigen Post gerne annehmen. Das hat nicht unbedingt was mit Ziegen, dafür aber mit genome engineering zu tun.
Kurz erklärt, geht es darin um die gezielte Einbringung von gewünschten genetischen Veränderungen in Zielzellen/Tiere. Sowas nennt man dann Knock-out bzw. Knock-in Zellen/Tiere. In den letzten Jahren ist das durch die Entwicklung ‚molekularer DNA-Scheren‘ erheblich erleichtert worden. Das sind Proteine, die DNA gezielt an einer ganz bestimmten Stelle schneiden können. Dieses Öffnen der DNA erleichtert es dann neue Sequenzen an der Stelle des Schnitts einzufügen. Zum Einbringen neuer Sequenzen sind außerdem sogenannte Homologiearme erforderlich. Das sind nichts weiter als die flankierenden Sequenzen an der Insertionsstelle. Die Zelle selbst baut dann über einen Prozess der ‚homologe Rekombination‘ genannt wird, die fremde DNA eigenständig in ihr Genom ein. Um das etwas zu verdeutlichen, hier ein Bild dazu.
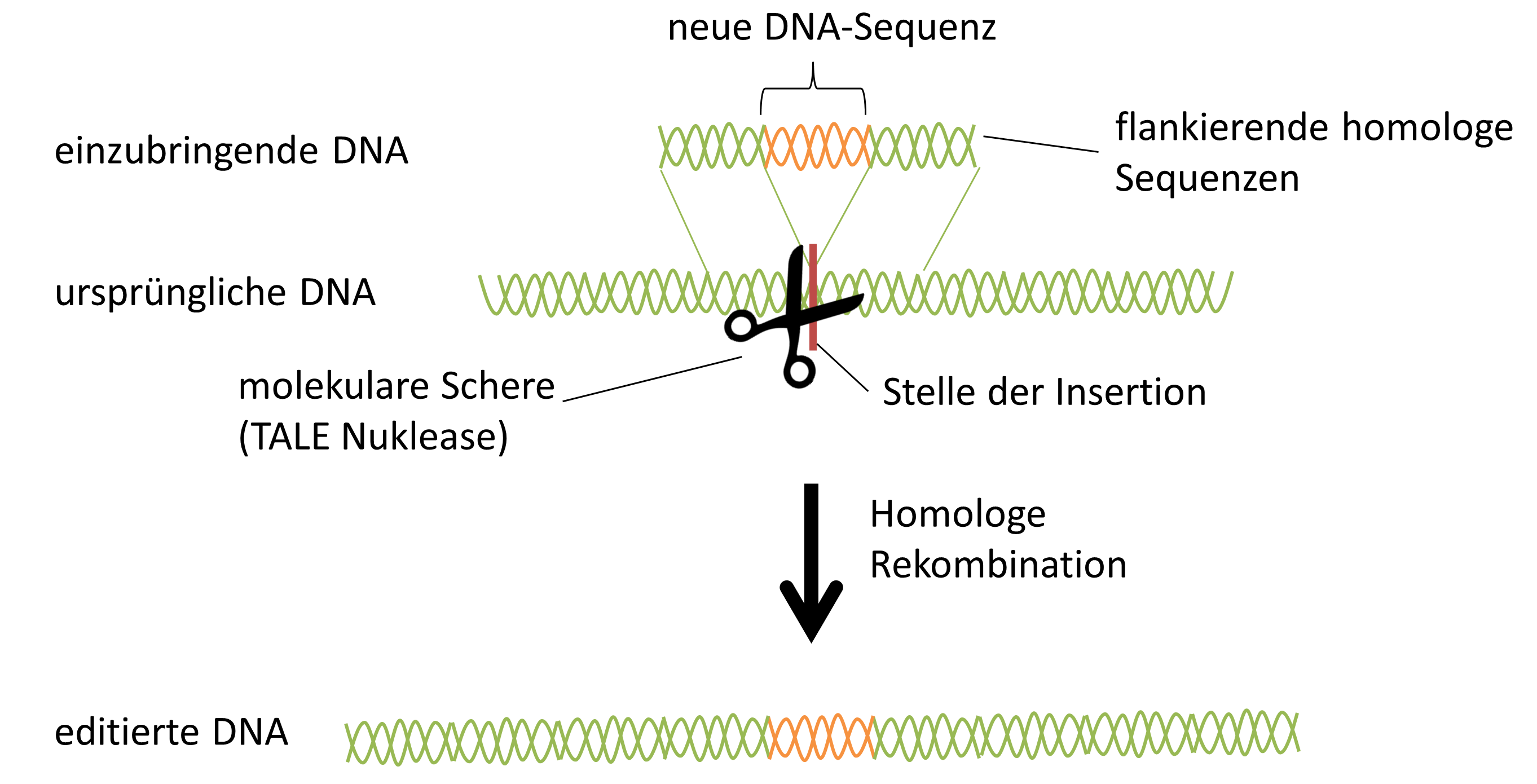 Die ursprüngliche DNA Sequenz ist in grün dargestellt, die neue Sequenz in orange. Um diese neue Sequenz über den zelleigenen Prozess der homologen Rekombination einzubringen, werden die flankierenden (oben, grün) Sequenzen der Insertionsstelle benötigt, die sich auch exakt so auf der ursprünglichen DNA befinden. Der Einbau kann spontan ablaufen, allerdings mit sehr geringer Effizienz (unter 1%). Wenn man jedoch eine molekulare Schere verwendet, die die DNA an dieser Stelle vorher öffnet, findet dieser Prozess mit einer sehr viel höheren Effizienz statt, wie ihr anhand der Veröffentlichung die ich gleich vorstellen werde, sehen könnt. Die Zelle ist dann einfach darum bemüht diesen DNA-Schaden zu beheben und eine Möglichkeit ist eben die homologe Rekombination.
Die ursprüngliche DNA Sequenz ist in grün dargestellt, die neue Sequenz in orange. Um diese neue Sequenz über den zelleigenen Prozess der homologen Rekombination einzubringen, werden die flankierenden (oben, grün) Sequenzen der Insertionsstelle benötigt, die sich auch exakt so auf der ursprünglichen DNA befinden. Der Einbau kann spontan ablaufen, allerdings mit sehr geringer Effizienz (unter 1%). Wenn man jedoch eine molekulare Schere verwendet, die die DNA an dieser Stelle vorher öffnet, findet dieser Prozess mit einer sehr viel höheren Effizienz statt, wie ihr anhand der Veröffentlichung die ich gleich vorstellen werde, sehen könnt. Die Zelle ist dann einfach darum bemüht diesen DNA-Schaden zu beheben und eine Möglichkeit ist eben die homologe Rekombination.
Die chinesische Gruppe wollte mit diesem System also genetisch veränderte Tiere erzeugen, die kein BLG mehr produzieren, sondern nach Möglichkeit humanes Lactoferrin. Auch in diesem Fall dürfen Tierversuche nicht einfach ohne Vorarbeiten in Zellkultursystemen durchgeführt werden. Zu diesem Zweck wurden zunächst molekulare Scheren (TALE Nukleasen) generiert, die spezifisch zwei Bereiche der genomischen Sequenz des BLG-Gens schneiden. Stattdessen sollte an dieser Stelle ein Selektionsmarker, eine Antibiotikaresistenz, eingefügt werden. Nachdem sie die verwendeten Homologiearme von anfänglich 1.000 bp auf über 5.000 bp homologe Sequenzen erweitert hatten, war dies dann auch möglich. Dazu werden die TALE Nukleasen und die neue DNA unter Verwendung elektrischer Impulse gemeinsam in die Zellen eingebracht. Einige Zellen bauen tatsächlich die neue DNA-Sequenz über homologe Rekombination ein und sind dann resistent gegen das gewünschte Antibiotikum. Diese Zellen lassen sich selektionieren, indem man ganz simpel das entsprechende Antibiotikum mit ins Nährmedium gibt und somit alle nicht-editierten Zellen absterben. Was dann übrig bleibt, bezeichnen wir als ‚Klon‘. Diese Antibiotika-resistenten Klone kann man dann einzeln kultivieren und untersuchen, ob die neue DNA tatsächlich integriert wurde, oder ob die Antibiotika-Resistenz nicht doch anderweitig zustande kommt (das ist gar nicht mal so selten). Hier wurden über 500 Klone untersucht, von denen ~ 18% auch die korrekte Editierung in sich trugen. Und das ist dann schon deutlich effizienter als die unter 1%, die man mit spontaner homologer Rekombination erreicht. In einer zweiten homologen Rekombination wurde diese Sequenz dann erfolgreich durch die des humanen Lactoferrins ausgetauscht.
Da das also in der Petrischale so wunderbar funktioniert hat, durfte dann auch ins Tiermodell gegangen werden. Hierfür wurde etwas verwendet, was man als SCNT (Somatischer Zellkerntransfer) bezeichnet. Wie der Name vermuten lässt, wird dabei der Zellkern einer somatischer Zelle (normale Körperzellen, z.B. Hautzellen) transferiert, und zwar in eine vorher entkernte Eizelle. Also: der Zellkern der Eizelle wird entnommen, dadurch verliert die Zelle quasi ihre genetische Identität. Dann kann man einen anderen Zellkern einsetzen, dessen DNA vorher meistens verändert worden ist um dann so genetisch veränderte Tiere erzeugen zu können. Das ist das gleiche Verfahren, was damals bei ‚Klonschaf‘ Dolly angewandt worden ist. Vielleicht erinnert sich der eine oder andere noch. So ein Bild wie unten, wird wohl jeder schon einmal gesehen haben. Hier, findet ihr außerdem ein Video dazu, wie das Ganze abläuft.
Ganz so einfach ist das natürlich nicht. Viele Zellen werden nach so einer Behandlung nicht normal weiter wachsen können. Durch Stimulation, elektrisch und chemisch, können die Zellen aber dazu angeregt werden sich wieder normal zu teilen. Die so behandelten Zellen werden dann erst einmal für mehrere Tage im Inkubator gehalten, bis man sicher sein kann, dass die Zellen normal weiter wachsen. Im Prinzip kultiviert man zu diesem Zeitpunkt bereits einen Embryo, der sich dann allerdings meistens erst im Zweizellstadium befindet. Erst nach dieser Überprüfung werden die Zellen in Muttertiere implantiert, in denen dann hoffentlich Nachwuchs heranwächst. So auch hier geschehen. Von 70 implantierten Zellkernen wurden zehn Klontiere erhalten.
Eine weitere Schwierigkeit beim genome editing ist es, tatsächlich auch beide Allele zu treffen. Wir Menschen haben ja bekannterweise einen doppelten Chromosomensatz. Das heißt im Idealfall, dass jedes Gen auf jedem Chromosom zweimal vorhanden ist. Die beiden Gene werden dann als Allele bezeichnet, da beide Gene in unterschiedlichen Ausführungen vorkommen können, die z.B. zu unterschiedlicher Aktivität eines bestimmten Enzyms führen können. Wenn man ein Gen also komplett ausschalten möchte, muss man folgerichtig auch beide Chromosomen (bzw. beide Allele des Gens) erfolgreich editieren. Die in diesem Experiment ausgetauschten Zellkerne waren anscheinend alle nur in einem Allel editiert, was eine zweite Runde erforderlich machte. Drei der insgesamt zehn erhaltenen Tiere trugen das Lactoferrin in sich, in den restlichen sieben Tieren wurde lediglich das BLG entfernt. Für die zweite Editierungsrunde wurde direkt von den Tieren genomische DNA entnommen, diese genau wie vorher editiert, so dass biallelische knock-out bzw. Lactoferrin-Knock-in Tiere erhalten wurden.
Insgesamt ist das ein Prozess der sehr viel Zeit in Anspruch nimmt und extrem arbeitsaufwändig ist. Insegsamt wurden 980 Klone in der Petrischale erzeugt und überprüft. In lediglich 163 Klonen ist das Gen BLG auch tatsächlich editiert worden. 136 Klone zeigten normales Wachstum, die restlichen Klone waren nicht in der Lage sich weiter zu teilen und wurden verworfen. Zehn verschiedene Klone wurden dann auf ihre Fähigkeit getestet sich nach erfolgten SCNT weiter zu teilen. Daraus resultieren dann besagte zehn Tiere und nach der zweiten Editierungsrunde waren es nur noch fünf geklonte Ziegen, drei Knock-out Tiere und zwei Tiere mit Lactoferrin Knock-in. Ohne die Möglichkeit die DNA mit den TALE Nukleasen aufzuschneiden, hätte es noch wesentlich länger gedauert die große Anzahl an Klonen im ersten Schritt zu erzeugen. Die Erzeugung genetisch veränderter Tiere für die Forschung wurde durch deren Entdeckung also erheblich erleichtert.
Aber eigentlich ging es ja um Ziegenmilch. Diese wurde dann auch entsprechend untersucht und rausgekommen ist dabei folgendes:
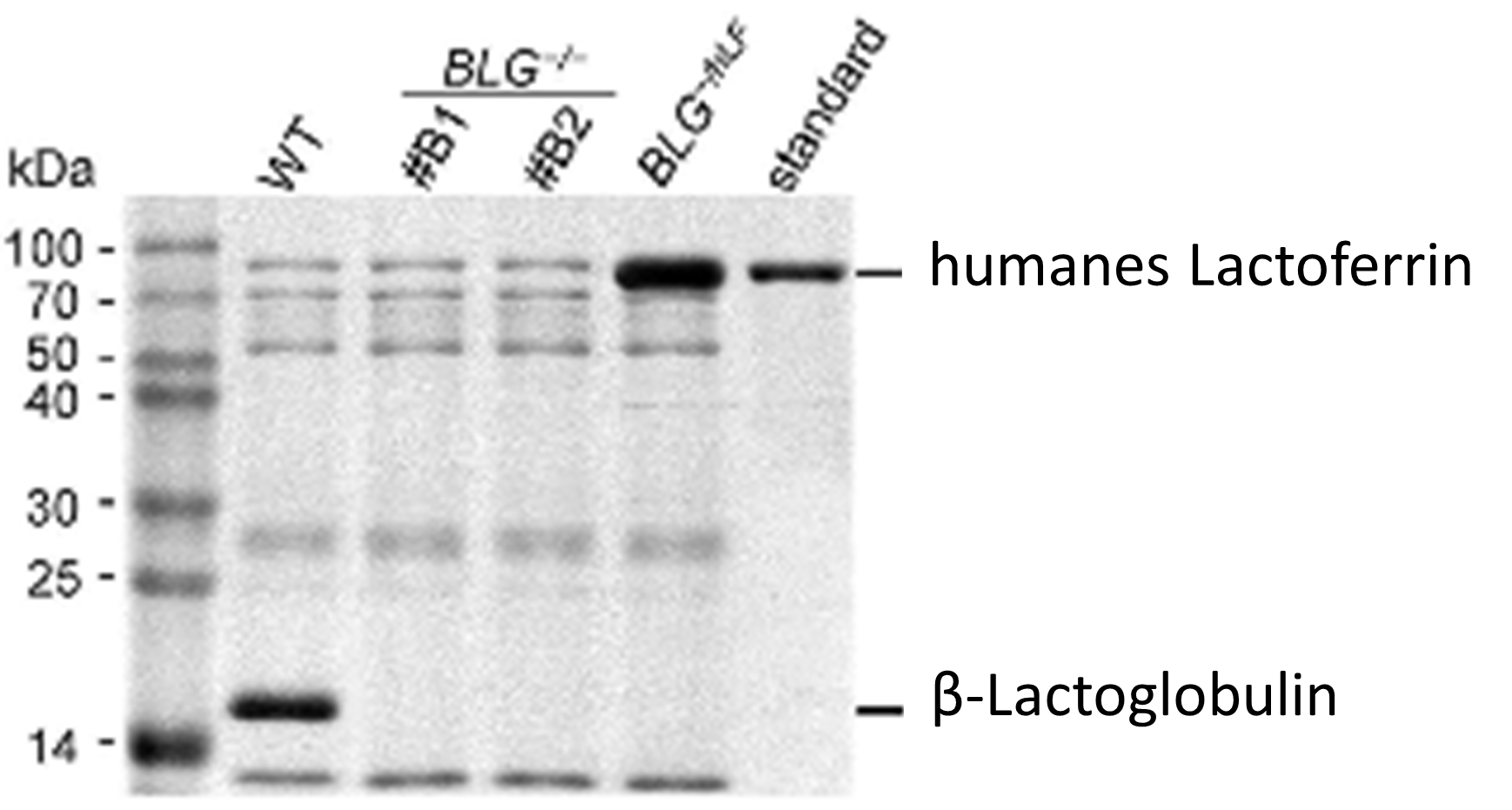
Das Bild oben zeigt einen sogenannten Westernblot. Dabei werden Proteine aus Zellen extrahiert und diese dann mit mehr oder weniger spezifischen Antikörpern für ein oder mehrere Protein behandelt. Wenn das gesuchte Protein vorhanden ist, bindet sein Antikörper. Diese Bindung kann dann angefärbt werden und wird als schwarze Bande sichtbar. Auf der linken Seite ist ein Größenstandard angegeben, mit dem sich überprüfen lässt, ob sich die beobachteten Banden auch tatsächlich dort befinden, wo das Protein zu erwarten ist oder ob die Antikörper unspezifisch etwas anderes gebunden haben. Auf der linken Seite ist markiert wo sich BLG bzw. Lactoferrin befinden sollten. Und tatsächlich die normalen Wildtyp (WT) Ziegen bilden viel BLG, aber kein humanes Lactoferrin. Die beiden untersuchten BLG Knock-out Tiere (#B1 und #B2) bilden weder BLG, noch Lactoferrin und die Lactoferrin Knock-in Tiere sind dann schließlich in der Lage humanes Lactoferrin zu bilden. Die ganze Arbeit war also nicht umsonst, am Ende ist die Ziegenmilch tatsächlich BLG frei.
Es gibt Leute, die werden sicherlich Bedenken haben, solche „Genmilch“ zu sich zu nehmen. Die haben dann aber wahrscheinlich auch keine Milchallergie. Auch ohne Milchallergie würde ich die Milch jedenfalls trinken, wobei der Geschmack von Ziegenmilch ja eher streng sein soll.
Merken


Danke für die detaillierte Darstellung. Sie zeigt mir unter anderem, dass heutige Verfahren des Genome Editing bei aller Präzision dennoch nicht bei 100% aller behandelten Zellen funktionieren. Mit entsprechender Nachkontrolle und Geduld erhält man aber schliesslich einige Tiere oder Pflanzen mit genau den gewüschten eingefügten oder gelöschten Genen an genau der anvisierten Stelle. Um modifizierte Arten zu “züchten” genügt die so erreichte Präzision sicher, denn beim hineinzüchten von neuen Merkmalem braucht es mit konventionellen Metoden noch viel mehr Geduld. Nach einigen Anläufen erreicht man mit dem Genome Editing also eine hochpräzise genetische Modifikation, die auch weitervererbt wird.
Hallo Martin,
mit so einer hohen Präzision rechnet eigentlich auch niemand. Das fängt schon damit an, das dass Einbringen der Nukleasen/Homologievektoren wohl eher zu im Schnitt 80% erfolgt und dann müssen auch noch verschiedene (meist 3) Vektoren gleichzeitig in eine Zelle eingebracht werden. Alleine von diesen Gesichtspunkten her sind 100% Präzision also extrem unwahrscheinlich und da ist auf biologischer Ebene noch gar nichts passiert.
Es ist aber genau wie du sagst. Diese neuen Nukleasen können zielgerichtet eingesetzt werden. Konventionelle Züchtung wie das bestrahlen mit UV-Licht verursacht dagegen multiple Mutationen die durch viel Probieren den gewünschten Genotyp erzeugt, meistens ohne wirklich zu wissen, welche genomischen Loci denn alle betroffen sind.
Besten Dank für die Bestätigung des kleinen oder mittelgrossen Genome-Editing yields (yield wie man bei der Herstellung von integrierten Schaltkreisen oder beim Ertrag einer angepflanzten Fläche sagen würde). Das bedeutet wohl, dass mit heutigen Genome-Editing Methoden Dinge wie die Korrektur von Gendefekten an Embryos noch nicht ohne weiteres realisiert werden können. Meine Phantasie ist da wohl mit mir durchgebrannt als ich mir vorstellte man könne schon bald sogar das Genom von Erwachsenen editieren und damit bestimmte Alterskrankheiten (oder auch andere Krankheiten) zum Verschwinden bringen.
Vor kurzem ist genau das mit humanen Embryonen aus einer Fertilitätsklinik in China versucht worden. Die Gruppe wollte das Gen was eine ß-Thalassämie hervorrufen kann, korrigieren. Allerdings wurden die Embryonen nicht implantiert. Hier ist der Link dazu: http://www.nature.com/news/chinese-scientists-genetically-modify-human-embryos-1.17378
War nicht besonders erfolgreich und das ganze Experiment wurde entsprechend abgebrochen.
Ja, die CRISPR/Cas9 Intervention an einem humanen Embryo durch eine chinesische Gruppe verlieft unbefriedigend mit mangelnder “Ausbeute”. Es hätte besser gemacht werden können meint der scilogs-Artikel CRISPR, Genome-Engineering und genmanipulierte Embryos: Spiel mit dem Erbgut?
Die Tendenz geht wohl schon in Richtung noch höhere Präzision und höhere Erfolgsraten.
Ein grosses Anwendungsgebiet des Genome Editing scheint die Agrikultur zu sein (Precision Genome Engineering and Agriculture: Opportunities and Regulatory Challenges), aber auch die Modellierung von menschlichen Krankheiten in Labortieren scheint ein Anwendungsfall zu sein.
Interessanterweise kann sogar das reine Ausschalten von Genen sich sehr günstig auf Kulturpflanzen auswirken, denn nicht alle Gene von Pflanzen dienen der Produktion von Nährstoffen, wie folgendes Zitat zeigt:
Ein wirklich spannendes Gebiet. Vielen Dank dass Sie darüber so hautnah und anschaulich berichten.