A wie Axolotl oder Salamander Superpower

“Es gab Tage, da wäre ich am liebsten gleich wieder nach Hause gegangen“. Und dabei muss man sie doch einfach mögen. Es gibt sie in verschiedenen Farben, von blassrosa bis schwarz, mit oder ohne Sommersprossen. Die Kiemenäste zu beiden Seiten des Kopfes tragen sie wie einen Heiligenschein, und sie sehen immer ein bisschen so aus, als würden sie einen anlächeln. Die Rede ist vom mexikanischen Schwanzlurch aus der Familie der Querzahnmolche, auch bekannt als Axolotl, was im Aztekischen soviel bedeutet wie Wassermonster. So weit, so niedlich.
Was den Axolotl für die Wissenschaft so interessant macht, ist die Tatsache, dass verlorene Gliedmaßen, Organe und sogar Teile des Gehirns oder des Herzens bei ihm nachwachsen, einfach so und notfalls mehrmals hintereinander, eine Eigenschaft, die sich der Mensch natürlich gerne abschauen würde. Doch dazu müsste man wissen, wie der Axolotl das anstellt.
Und genau da wird es kompliziert: Mit 32 Milliarden Basenpaaren ist das Genom, also der genetische Bauplan des Axolotl, zehn Mal so groß wie das des Menschen und das bislang umfangreichste Genom überhaupt, das wir kennen. Um es zu entschlüsseln, braucht es neben einem leistungsfähigen Computer ein gerüttelt Maß an Geduld, Einfallsreichtum und Frustrationstoleranz, was im eingangs zitierten Satz des ehemaligen HITS-Juniorgruppenleiters Siegfried Schloissnig bereits anklingt. Doch dazu später mehr.
Die Geschichte unseres ersten Forschungshighlights beginnt im Jahr 2013. Damals führte eine Reihe von glücklichen Zufällen den Bioinformatiker Schloissnig ans HITS, wo er die Juniorforschungsgruppe „Computational Biology“ gründete. Zur gleichen Zeit leitete die Biochemikerin Elly Tanaka am Max-Planck-Institut für molekulare Zellbiologie und Genetik (MPI-CBG) in Dresden eine Forschungsgruppe, die sich den Axolotl als Modellorganismus erwählt hatte.
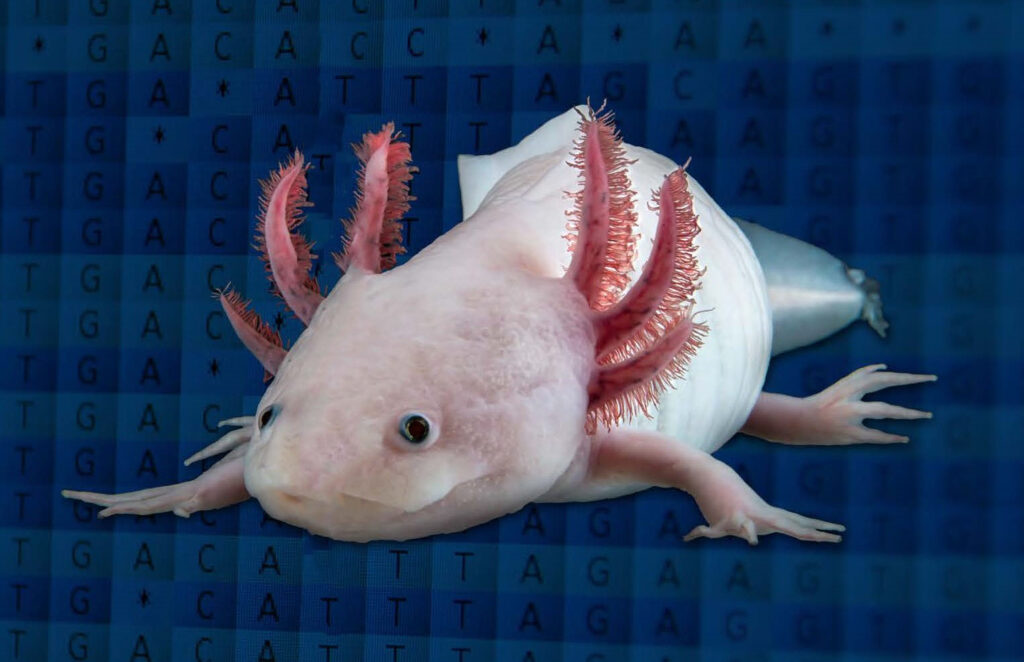
Sie und ihr Team hatten bereits Fragmente der Axolotl-DNA sequenziert, das heißt, sie hatten kleinere Abschnitte des genetischen Bauplans entschlüsselt – aber eben nur kleinere Abschnitte. Die Tatsache, dass sich das Axolotl-Genom durch eine hohe Anzahl von sich wiederholenden Teilen auszeichnet, machte das Ganze nicht gerade leichter. Für die Zusammensetzung dieser schier endlosen Anzahl von Fragmenten und die komplette Darstellung fehlten damals noch die nötigen Ressourcen, vor allem Zeit und die geeignete Software. Beides fanden Siegfried Schloissnig und sein Team am HITS.
Und so machten sie sich 2013 ans Werk, um eine Software zu entwickeln, mit der die gigantische Aufgabe gelöst werden sollte. Doch wie geht man ein solches Projekt an? Zunächst einmal, indem man sich gründlich vorbereitet. Über ein Jahr lang stellte das Team am HITS gemeinsam mit Kollegen aus Dresden und Wien Kriterien auf, wie der neue Assembler – so nennt man diese Computerprogramme in der Bioinformatik – beschaffen sein müsste, um die schier endlose Abfolge von Basenpaaren des Genoms zu einem aussagekräftigen Ganzen zusammenzufügen.
„Dank unseres Stifters Klaus Tschira hatten wir genügend Zeit und keinen Publikationsdruck. So konnten wir in dieser schönen Umgebung am HITS ungestört unserer Arbeit nachgehen“, erinnert sich Schloissnig.
Parallel dazu wurden am Max-Planck-Institut für molekulare Zellbiologie und Genetik in Dresden und am Forschungsinstitut für Molekulare Pathologie in Wien weitere Teile des Axolotl-Genoms sequenziert.
Man kann sich das in etwa wie bei einem gigantischen Puzzlespiel vorstellen, allerdings mit mehr als den handelsüblichen Teilen. Die Sequenzierer, also die Maschinen, die aus dem organischen Material die Abfolge der Basenpaare in der DNA bestimmen, liefern lediglich die Puzzleteile. Aufgabe des Assemblers ist es, sie so zusammenzusetzen, dass daraus ein möglichst vollständiges und sinnvolles Gesamtbild entsteht.
Ein glücklicher Umstand war, dass zu dieser Zeit erhebliche Fortschritte auf dem Gebiet der Sequenzierungstechnologie gemacht wurden, das heißt, die Teile, die Siegfried Schloissnig und seinem Team zur Verfügung standen, wurden immerhin zunehmend größer, was ihre Zuordnung und damit die Zusammensetzung wesentlich erleichterte. Am Ende waren es aber immer noch über 72 Millionen Puzzleteile, was eine ungefähre Vorstellung davon vermittelt, mit welchen Größenordnungen es die WissenschaftlerInnen zu tun hatten.
Was folgte, waren vier Jahre Programmierarbeit für eine Software, deren Struktur von Grund auf neu geschaffen werden musste. Ein Vorteil dabei war, dass man von Anfang an zielgerichtet arbeiten und bei der Programmierung alle überflüssigen Komponenten gleich weglassen konnte, was unter anderem Rechenleistung einsparte. Einen Hinweis auf die schwierigeren Phasen des Projekts und den Rückschlägen gibt das Zitat von Siegfried Schloissnig vom Anfang des Textes.
Ungefähr 30 Wochen, 300.000 CPU-Stunden und ein paar schlaflose Nächte später war es dann schließlich soweit: Die erste vollständige Entschlüsselung des Axolotl-Genoms war erfolgreich abgeschlossen, oder, anders gesagt, der genetische Bauplan lag vor. Und siehe da, die Analyse des Genoms offenbarte einige Merkmale, die auf die Einzigartigkeit des freundlich dreinblickenden Schwanzlurchs hinweisen: Einerseits fanden die WissenschaftlerInnen mehrere Gene, die nur beim Axolotl und anderen Amphibienarten vorkommen und bei der Regenerierung von Gewebe eine aktive Rolle spielen. Andererseits fehlt ein wichtiges und weit verbreitetes Entwicklungsgen namens PAX3 beim Axolotl vollständig. Dessen Funktion übernimmt stattdessen ein verwandtes Gen namens PAX7. Beide Gene spielen eine Schlüsselrolle bei der Entwicklung von Muskeln und Nerven.
„Jetzt wo wir das Puzzle erfolgreich zusammengesetzt haben, können die Kolleginnen und Kollegen im Labor mit dem Axolotl-Genom wirklich etwas anfangen und das Ratespiel hat ein Ende“, fasst Schloissnig das wichtigste Ergebnis des Projekts zusammen.
Krönender Abschluss ihrer Arbeit war die Veröffentlichung der Studie samt Titelbild in „nature“, einer der renommiertesten Fachzeitschriften, in der WissenschaftlerInnen publizieren können.
Ebenfalls programmgemäß löste sich die Juniorforschungsgruppe „Computational Biology“ 2018 nach fünf Jahren auf, und die fünf Gruppenmitglieder arbeiten heute an verschiedenen Instituten in Österreich und Deutschland. Siegfried Schloissnig allerdings ist den Molchen treu geblieben. Er forscht am Institut für Molekulare Pathologie in Wien, wo das Labor von Elly Tanaka eine der weltweit größten Axolotl-Kolonien beherbergt – wie gesagt: man muss sie einfach mögen.
Falls Sie mehr über das Forschungsprojekt wissen möchten, empfehlen wir folgende Seiten:


Somatisches Gen-Editing mit CRISPR an der Amputationsstelle sollte es irgendwann ermöglichen, die jetzt nur beim Axolotl beobachtete Regenerationsfähigkeit von Gliedmassen und Organen auf andere, dazu nicht fähige Tierarten zu übertragen
So einfach mindestens stelle ich mir das als Laie vor. Es könnte natürlich ohne weiteres sein, dass die Regeneration einer Gliedmasse oder eines Organs den ganzen Körper fordert und es nicht genügt nur gerade die Regenerationsstelle aufzupeppen. Aber das Naheliegendste ist schon, dass das Geheimnis der Regeneration in den Genen der Wunde liegt und die Gene in nicht betroffenen Körperstellen keine Rolle spielen.
Mit der Genomsequenzierung des Axolotls ist man nun in meiner Einschätzung erst am Anfang eines jahre- bis jahrzentelangen Prozesses bis es schliesslich vielleicht gelingt die Regenerationsfähigkeit des Axolotls auf andere Species (inklusive Mensch) zu übertragen.
Es scheint, Forschung und Technologie kommen nie an ihr Ende. Wer von der baldigen Singularität ( The Singularity Is Near: When Humans Transcend Biology ) oder vom Zeitalter des Homo Deus spricht, der kann damit nicht das 21. Jahrhundert meinen, sondern allenfalls das 23. Jahrhundert – also eine Zeit, die von heute so weit entfernt ist wie das 18. Jahrhundert, ein Jahrhundert in dem man noch nichts wusste von DNA und Computern.
The protein tyrosine phosphatase 1B inhibitor MSI-1436 stimulates regeneration of heart and multiple other tissues:
https://www.nature.com/articles/s41536-017-0008-1
Trodusquemine:
https://en.wikipedia.org/wiki/Trodusquemine
Die Regeneration durch eine hormonartige, per Infusion verabreichte Substanz, wäre natürlich am bequemsten. Die von Karl Bednarik verlinkte Studie The protein tyrosine phosphatase 1B inhibitor MSI-1436 stimulates regeneration of heart and multiple other tissues berichtet genau über eine solche Substanz, die bei Zebrafischen und Mäusen in den Bauchraum gespritzt wurde und die dann beim Zebrafisch das Ausheilen/Regenerieren einer amputierten Schwanzflosse anstiess und die bei Mäusen die Ausheilung einer Herzinfarktwunde verbesserte (Vermehrung der Kardiomyozyten) und damit die Überlebensrate nach Herzinfarkt erhöhte. Hier ein Ausschitt aus der Kurzzusammenfassung des Artikels (übersetzt von DeepL):
BEURTEILUNG: Nicht nur Fische und Amphibien, auch Säugetiere verfügen über eine natürliche Regenerationsfähigkeit. So wachsen bei Kleinkindern abgetrennte Fingerkuppen bekannterweise nach ohne eine sichtbare Spur zu hinterlassen. Bestimmte Substanzen könnten diese natürliche Fähigkeit auch beim Erwachsenen reaktivieren. Die Regeneration ganzer Gliedmassen aber scheint wirkliche dem Axolotl vorbehalten zu sein. Und sie bedarf wohl spezieller Gene und diese Gene sind heute nur beim Axolotl nachgewiesen.
Nachtrag:
Molekularer Jungbrunnen, MSI-1436:
https://www.spektrum.de/magazin/molekularer-jungbrunnen/1669380
Aus Sicht der technischen Informatik gibt es eine ganz banale Erklärung für die „ungewöhnlichen“ Eigenschaften des Axolotl.
Es liegt einfach daran, dass die Evolution bei der Entwicklung der Amphibien eher am Anfang ihrer Entwicklung stand und gewisse grundlegende Programm Konzepte für die von genetischen Programmen gesteuerten Prozesse noch nicht vorhanden waren.
Es gab, ganz einfach formuliert, noch keine „ausgefeilte Unterprogrammtechnik“.
„Gleiche Programmteile“ (DNA Code) konnten (noch) nicht oftmals „verwendet“ werden.
Bedeutet, wird ein Körperteil abgetrennt, wird der diesen „ersetzende“ zuständige Programmteil noch einmal „exprimiert“ und der Teil wächst ungestört nach, so wie ursprünglich. Es gibt sozusagen keine „störenden Sprunganweisungen im Hauptprogramm“, das Programm „läuft“ linear ab und das Teil kann nachwachsen.
Allerdings ist klar, dass bei dieser primitiven „Programmtechnik“ immens viel Programmcode notwendig ist, weil es keine Möglichkeit gibt, dass Programmteile einzeln selektiv oftmals „exprimiert“ (die Informatiker sagen “aufgerufen”) werden können.
So ist es ganz banal erklärbar, dass der Mensch viel weniger Programmcode (DNA) benötigt als der Axolotl, dennoch wesentlich komplexer funktioniert und leistungsfähiger ist.
Wollte man ein Nachwachsen von Körperteilen beim Menschen, müsste in die komplexe „Unterprogrammtechnik“ eingegriffen werden, was nicht so einfach scheint.
Auch die Evolution brauchte für die Entwicklung „ihrer systematischen Konzepte“ Zeit (und andere Ressourcen).
@Elektroniker: letztlich funktionieren auch mehrzellige Organismen dezentral und viele Zellen verfügen über das komplette Programm um sich zu duplizieren und zu regenerieren. Eventuell benötigt es Signale von aussen um die „Exprimation“ anzustossen. Falls aber bei Nicht-Axolotl-Organismen In den entscheidenden Zellen Programmteile fehlen könnten sie durch Gen-Editing (CRISPR) hinzugefügt werden. Eventuell genügt eine Reprogrammierung an der Amputationsstelle um Zellen zu schaffen die zur Regeneration fähig sind.
Mir geht es also darum, die Regeneration von Gliedmassen und Organen beim Menschen wieder zu ermöglichen. Eine Reprogrammierung durch Gen-Editing mittels CRISPR erscheint mir vielversprechend.
@ Martin Holzherr
Bei der Regeneration von Gliedmaßen geht es um die Prozesse im Zusammenhang mit der Genexpression. Einerseits um den DNA Code, der zunächst einmal die den Code entsprechende RNA generiert. Und einen Mechanismus der die „Aktivierung“ der jeweils relevanten DNA Codeelemente bewirkt, so etwas wie einen dynamischen Exprimierungsmechanismus.
Ich vermute, dass sich auf der Stammzellen DNA (der höchsten Hierarchieebene) so etwas wie besondere „Markierungen“ befinden, die es ermöglichen dass der Exprimierungsprozess sozusagen von Markierung zur nächsten Markierung „springt“ und ein Programmteil nach dem nächsten „abgearbeitet“ und auch neue DNA erzeugt wird.
Dabei entstehen bei neueren Systemen ebenfalls markierte DNA Abschnitte ohne „globalen“ Stammzellencharakter, es werden also nur mehr ?wenige? allenfalls nur mehr ein einziger Abschnitt „abgearbeitet“ und es entsteht in diesem Fall immer der gleiche Zelltyp.
Seit einiger Zeit steht fest dass es mindestens 2 Hierarchieebenen an „DNA Typen“ gibt. Es wäre aber denkmöglich dass es mehrere derartige Ebenen gibt? Frühe primitive Systeme dürften nur über die Stammzellenhierarchie verfügen.
Dieser Mechanismus würde ungefähr der Unterprogrammtechnik entsprechen. Jede neue Zelle bekommt bei der Zellteilung eine entsprechend relevant markierte neue DNA die die jeweiligen Zellbildungsprozesse auch abhängig von den vorhandenen Ressourcen steuert.
Angeblich wachsen auch bei Kleinkinder z.B. abgetrennte Fingerkuppen nach. Dies wäre damit zu erklären, dass einfach der Zellbildungsprozess (nur) auf der untersten Hierarchie wieder aufgenommen wird und wie beim Axolotl die Fingerkuppe wieder nachwächst.
Wird aber mehr als nur ein kleiner Teil der Kuppe abgetrennt, so müsste ganz exakt die genau zum Abtrennungsmuster passende Unterprogrammhierarchie erzeugt und aktiviert werden. Diese Mechanismen versteht man aber nur unzureichend. Man kann zwar Quadratmeter von Haut erzeugen, Spezialfirmen können auch schon Kunstfleisch erzeugen. Aber so richtig vielfältige Strukturen mit Adern, Sehnen, Knochen … zu bauen, dafür dürften die Grundlagenkenntnisse noch nicht reichen. Die verteilten Steuerungsprozesse sind sehr komplex, weil ja auch eine zeitliche Koordinierung der einzelnen Prozesse erforderlich ist. In diesem Zusammenhang vermute ich, dass die nicht codierende DNA Einfluss darauf nimmt.
Darüber erfährt man kaum etwas. Ich vermute dass Fachfirmen aus Patent rechtlichen Gründen Informationen zurückhalten und das Personal unter Verschwiegenheit steht.
Ich kann es nur aus Sicht der Elektronik/Informatik sehen, da auch dort aus linear übertragener Software (wie DNA) komplexe und lokal verteilte Prozesse z.B. für Produktionsanlagen gesteuert werden müssen. Nahe liegender Weise gibt es Ähnlichkeiten mit genetisch gesteuerten Prozessen der Biologie.
Ich bin kein Biologe. Kann nur versuchen für allgemein bekannte Probleme der Biologie Lösungsansätze (systematische Funktionsprinzipien) aus der Elektronik/Informatik vorzuschlagen.
Ähnlichkeiten gibt es bei den baumartigen neuronalen Strukturen und bei den linearen Strukturen der Gentechnik.
Es wird im Prinzip „lineare“ Information auf DNA Strukturen so umgesetzt, dass letztlich die bekannten Zellstrukturen generiert werden. Im Gehirn sind es die vorbereiteten neuronalen Strukturen die vom Input, auch im Zusammenwirken mit dem eigenen Output (Rückkoppelung) „programmiert“ werden.
Im Prinzip entspricht die DNA einen linearen Programmspeicher der die Information zur Steuerung der Prozesse für die Erzeugung der jeweilige RNA enthält. Dann ist auch noch die Adressierung des jeweiligen Abschnittes auf der DNA bedeutend, dies erledigt in der Elektronik der Programmspeicher.
Eine Reprogrammierung durch Gen-Editing mittels CRISPR erscheint denkmöglich, wenn man künstlich genau die zur Erzeugung der fehlenden Zellen erforderliche DNA erzeugt. Derzeit dürfte man noch zu wenig über die grundlegenden Steuerungsprozesse der Genexpression wissen.
@Elektroniker: Die Regeneration von Gliedmassen und Organen kann letztlich als Reaktivierung eines Entwicklungsprozesses aufgefasst werden, der in der Embryonal-/Fötalphase bereits einmal ablief. Um das im Erwachsenenorganismus anzustossen müssen gewisse Zellen in den Embryonalzustand zurückversetzt werden.
Wenn man das auf programmierbare Systeme überträgt, so muss man früher aktive Programme reaktivieren – dies aber mit der Hardware von heute, die nicht mehr die gleiche wie die ursprüngliche ist.
@Elektroniker (Zitat):
In Regeneration and repair of human digits and limbs: fact and fiction liest man dazu (übersetzt von DeepL):
FAZIT: Verletzte Fingerkuppen regenerieren bei Kleinkindern häufig, bei älteren Kindern seltener und kaum noch bei Erwachsenen. Vielleicht liegt das auch an Regenerationshemmnissen beim Erwachsenen. Es könnte also sein, dass man nur Regenerationsbremsen / Regenerationsstopper entfernen muss um die Regeneration wieder zu ermöglichen.
Analogon: die Krebsimmuntherapie erwuchs gerade aus der Beobachtung, dass das Immunsystem fast immer auf Krebs reagiert, aber dass seine Aktion durch die Krebszellen inhibiert/gestoppt wird. Bei der Therapie mit Checkpoint-Inhibitoren beispielsweise gibt es beim Krebs- Patienten inhibitorische Immun-Checkpoints, welche verhindern, dass der Krebs bekämpft werden kann. Wenn diese Bremse gelöst wird, indem man den Immun-Checkpoint hemmt (typischerweise mit monoklonalen Antikörpern), dann kommt das Immunsystem wieder zum Zug.
Wer weiss: Vielleicht hat ja selbst der Erwachsene noch eine gute Regenerationsfähigkeit für vieles und sie ist nur ausgebremst. Dann müsste man nur die Bremsen lösen um beispielsweise einen Finger nachwachsen zu lassen.
Eine Lebensmittelfarbe könnte nach Wirbelsäulenverletzungen das Schlimmste verhindern:
https://www.spektrum.de/news/blau-ist-die-heilung/1003001
Systemic administration of an antagonist of the ATP-sensitive receptor P2X7 improves recovery after spinal cord injury:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2718350/
Das Heidelberger Institut für Theoretische Studien (HITS) beschäftigt sich eigentlich gar nicht mit dem biologischen Phänomen der Regeneration oder mit anderen angewandten Wissenschaften, sondern es ist ein Stätte wo theoretische Studien (fast) alles angehen und hinterleuchten. Mit der Sequenzierung des Axolotl-Genoms ist seine Arbeit (vorerst) getan. Und in dieser Arbeit ging es vor allem um die Methoden mit denen man komplexe (beispielsweise hochrepetitive) Genome entschlüsseln kann.
Wir sprechen ja immer von empirischen Wissenschaften. Doch diese empirischen Wissenschaften beziehen ihre Stärke zum grossen Teil aus mathematisch fundierten Theorien. Diese Theorien sind mit den heutigen informatischen Mitteln so stark geworden, dass sie eine parallele Quasi-Realität schaffen. Irgendwann kann man im (digitalen) Labor die ganze Welt erschaffen – so scheint es mir.
@ Martin Holzherr
Als Reaktivierung eines Entwicklungsprozesses, so kann man es sicherlich sehen.
Es dürfte darauf ankommen die DNA in „Grenzzellen“ (an der Abtrennungsstelle) in den genau „passenden Status“ zu versetzen um z.B. einen Finger nachwachsen zu lassen. Möglichst auch noch schneller als in der Embryonalphase, damit es nicht z.B. 20 Jahre dauert bis der Finger voll nachgewachsen ist.
Wenn man die genetische Prozesssteuerung möglichst voll versteht, so könnte man das erforderliche DNA Steuerungsprogramm (sozusagen mit den „Unterprogrammen“ aber ohne eingebaute „Bremsen“ (vermutlich Teile der nicht Eiweiße kodierenden DNA)) neu „designen“, so dass ein gut passender Finger neu entstehen könnte.
Salopp gesagt, man könnte die Original Stammzellen DNA so „zurechtschneiden“ und die passenden „Unterprogramm Markierungen“ einfügen, dass sozusagen die genau passende (DNA) Steuerungssoftware entsteht.
In der Technik ist derartiges in technischen Systemen üblich um auf das Prozessgeschehen (verbessernden) Einfluss zu nehmen. Um dies möglichst effizient zu ermöglichen, wurden Programmiertools entwickelt, die es ermöglichen hoch systematisch und rationell Software neu zu entwickeln, bestehende Software an neue Gegebenheiten anzupassen, bzw. zu analysieren (Software Reengineering).
Es gibt heutzutage „Emulatorsoftware“ die es ermöglicht, auf einem neuen Computer einen längst verschrotteten alten Computer zu „emulieren“, also z.B. uralte (sehr teure) Software nutzen zu können.
Danke für diesen interessanten Einblick in die Mühen der Genforschung.
Sie schreiben:
Ja, aber vielleicht sollte man hinzufügen, dass die Menge der Protein-codierenden Gene durchaus in einem Bereich liegt, wie wir es von den Wirbeltieren her kennen (ca. 23.000).
Woraus man folgern kann, dass sich bei dieser Spezies im Laufe der Evolution mehr „Datenmüll“ angesammelt hat als bei jeder anderen Tierart (soweit untersucht).