Nach zehn Jahren noch immer Lücken im menschlichen Genom
BLOG: Die Sankore Schriften

Das Humangenomprojekt (HGP) wurde im Herbst 1990 mit dem Ziel gestartet, das Genom des Menschen vollständig zu entschlüsseln. Im Juni 2000 wurde vom damaligen US-Präsidenten Bill Clinton unter Pauken und Trompeten die Sequenzierung des menschlichen Genoms in seiner groben Fassung verkündet.
Im April 2003 wurde das Human Genome Projekt offiziell als beendet erklärt. Die Sequenzierung war nach der technischen Definition des Projektteams beendet. Das hieß weniger als 1 Fehler pro 10 000 Nukleotide und 95% der proteinkodierenden DNA sequenziert.
Heute enthält die Sequenz des menschlichen Genoms (fast 3 Milliarden Basenpaare) immer noch ca. 350 Lücken, also Bereiche die nicht sequenziert sind. Sie sind unterschiedlich groß und reichen von 700 Basenpaaren bis zu 300 Millionen Basenpaaren. Die meisten Lücken befinden sich in den Bereichen der Chromosomen, die man Centromere und Telomere nennt. Sie sind in der folgenden Abbildung grün markiert. (Für eine größere Ansicht auf das Bild klicken.)

Abb.: Unsequenzierte DNA-Bereiche (Grün) auf den menschlichen Chromosomen
Das Centromer bzw. die Spindelfaseransatzstelle ist die primäre Einschnürungsstelle eines Chromosoms. Das Centromer teilt das Chromosom in zwei oft unterschiedlich lange Schenkel oder Arme.
Telomere sind die einzelsträngigen Enden der Chromosomen.
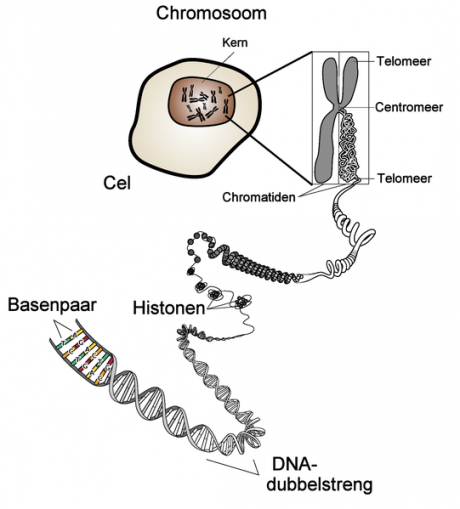
Abb.: Hierarchische Struktur der genetischen Information: Gen < DNA < Chromosom < Zellkern
Mehr zum Aufbau des menschlichen Genoms in meinem Artikel
Vergleich zwischen einer CD ROM und dem menschlichen Genom.
Literatur
The genome finishers, Elie Dolgin, (2009), Nature, Volume 462, S. 843-845.
Bildnachweis
Bild „Unsequenzierte DNA-Bereiche (Grün) auf den menschlichen Chromosomen“
Modifizierte Abbildung aus The genome finishers, Elie Dolgin, (2009), Nature, Volume 462, S. 843-845.
Quelle: Nature
Bild „Hierarchische Struktur der genetischen Information: Gen < DNA < Chromosom < Zellkern“
“Modell der Feinstruktur eines Chromosoms“
Quelle: Wikimedia

