Die Gene ALX1 und HMGA2 bestimmen Schnabelform- und größe in Darwinfinken

Am 27. Dezember 1831 beginnt Charles Darwin (22) seine Weltreise mit dem Forschungs-und Vermessungsschiff HMS1 Beagle. Die kleine Brigg startet von Plymouth in England und soll die Küsten Südamerikas vermessen und über Australien und Afrika wieder nach England zurückkehren. Die Reise dauert fünf Jahre, während der Darwin viele Tiere aus unterschiedlichen Regionen der Welt sammelt. Vom 15. September bis zum 20. Oktober 1835 hält sich die HMS Beagle vor Ecuador im Gebiet der Galápagosinseln auf. Unter den auf diesen Inseln geschossenen Vögeln, befinden sich 31 Exemplare der Galápagosfinken, die später Darwinfinken genannt werden.
Zurück in England geht Darwin ans Sortieren der Tiersammlungen, die er auf seiner Reise zusammengetragen hat und stößt immer wieder auf dieselbe Frage: Handelt es sich bei geringfügig unterschiedlich aussehenden Tieren nur um Abweichungen innerhalb einer Population oder um verschiedene Arten? Wodurch entsteht diese Art von Vielfältigkeit? Wie wird sie von Generation zu Generation aufrechterhalten? Mit diesem Rätsel schlug Darwin sich sein Leben lang herum, aber trotz aller Bemühungen fand er die Antwort nie.
Manchmal lassen sich diese Abweichungen in Klassen einteilen – das bezeichnen Biologen dann als Polymorphismus (griechisch ‚Vielgestaltigkeit‘). Wir kennen genetisch bedingte Polymorphismen von Mendels Experimenten mit Erbsen. Manche Erbsenpflanzen hatten weiße Blüten, manche rote, manche Erbsensamen waren grün, andere gelb, manche Erbsensamen waren rund, andere runzlig.
Darwin und Mendel waren Zeitgenossen, standen aber nicht in wissenschaftlichem Austausch miteinander. Darwins Buch “The Different Forms of Flowers on Plants of the Same Species”, hätte auch zu der Entdeckung führen können, die Mendel gemacht hatte. Aber Darwin hatte sein Augenmerk auf etwas anderes gerichtet: Quantitative Merkmalsvariation war das Herzstück von Darwins Evolutionstheorie und quantitative Variation ist der letzte Ort, an dem die Mendelsche Vererbung in reiner Form zu sehen wäre [1]. Zusätzlich war zunächst unklar, wie die natürliche Selektion eine kontinuierliche Veränderung eines quantitativen Merkmals wie z. B. die Größe einer Erbsenpflanze bewirken kann, wenn die Erbfaktoren laut Mendels – bei Polymorphismen entdeckten – Regeln diskrete Einheiten sind. Diesen scheinbaren Widerspruch konnte Ronald Fisher 1918 auflösen, indem er zeigte, dass durch das Zusammenwirken vieler Gene genau jene kontinuierlichen Veränderungen auftreten, die in der Natur beobachtet werden.
Heute ist die Genetik viel weiter als zu Mendels Zeiten und ein fester Bestandteil der Evolutionsforschung. Evolutionsgenetiker versuchen daher für Polymorphismen wie z. B. die Schnabelform und quantitative Merkmalsvariationen wie die Schnabelgröße, an denen die natürliche Selektion ansetzen kann, Gene zu finden.
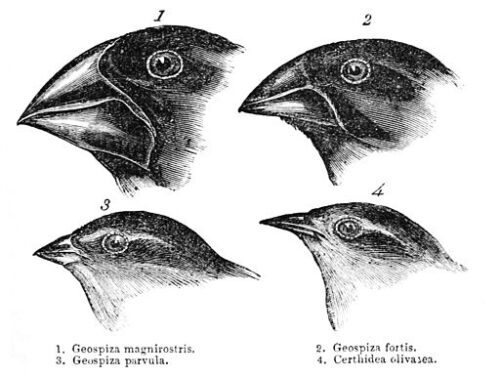
Bei Darwinfinkarten sind Schnabelform- und größe Adaptationen. In der Evolutionsbiologie meint Adaptation die genetisch bedingte Anpassung, die aufgrund von Mutationen und der natürlichen Selektion entstanden ist. Verantwortlich für die Schnabelform- und größe sind spezifische Zellen der Neuralleiste, die in der Embryonalentwicklung zum Schädel wandern. Unbekannt war bisher, welche Gene für die Form und Größe eines Schnabels eine Rolle spielen. Leif Andersson von der Uppsala Universität in Schweden und sein Forscherteam haben deshalb bei der Gattung Grundfinken (Geospiza) auf den Galápagosinseln nach Genen gesucht, die für die erbliche Variation der Schnabelform verantwortlich sind [2].
Dafür verglichen sie genomweit die DNA-Sequenzen von Grundfinken mit stumpfem Schnabel mit denen mit spitzem Schnabel. Die Wissenschaftler untersuchten drei Grundfinkarten und vier Grundfinkpopulationen:
- Großgrundfink (Geospiza magnirostris) mit stumpfem Schnabel auf der Insel Española
- Opuntien-Grundfink (Geospiza conirostris) mit stumpfem Schnabel auf der Insel Española
- Opuntien-Grundfink (Geospiza conirostris) mit spitzem Schnabel auf der Insel Genovesa
- Spitzschnabel-Grundfink (Geospiza difficilis) mit spitzem Schnabel auf der Insel Wolf
Die meisten Unterschiede in den DNA-Sequenzen fanden die Genetiker im Gen ALX1 (ALX homeobox 1). Das Gen ALX12 codiert für ein Protein, das für die normale Entwicklung des Schädels und Gesichts bei Wirbeltieren notwendig ist. Mutationen, die das Gen ALX1 inaktivieren, verursachen beim Menschen frontonasale Dysplasie, eine spezifische Störung der Embryonalentwicklung, die zu Missbildung der Nase, Stirn und Augen führt. Das Gen ALX1 ist vermutlich auch in den Zellen der Neuralleiste aktiv, die für die Bildung des Schnabels verantwortlich sind.
Das Protein ALX1 ist Transkriptionsfaktor. Transkriptionsfaktoren sind regulatorische Proteine, die spezifische Sequenzen in der DNA binden, um die Genexpression zu fördern oder zu unterdrücken. Daher können sie entweder Aktivatoren sein, die die Genexpression “einschalten”, oder Repressoren, die die Genexpression “ausschalten”. Insbesondere steuert der Transkriptionsfaktor ALX1 die Aktivität von Genen, die das Zellwachstum und die Zellteilung (Proliferation) und die Zellbewegung (Migration) regulieren. Diese Gene stellen während der Embryonalentwicklung sicher, dass Zellen zu bestimmten Zeiten wachsen und aufhören zu wachsen und dass sie im Körper richtig positioniert sind.
Die Genetiker fanden zwei Varianten, sogenannte Allele, des ALX1-Gens bei Grundfinken. Das Allel B (b = blunt) führt zu stumpfen Schnäbeln, das Allel P (p = pointed) zu spitzen. In den Körperzellen der Grundfinken gibt es die allermeisten Gene zweimal. Ein mütterliches Gen und ein väterliches Gen. Haben Mutter und Vater, das gleiche Allel (also die gleiche Variante eines Gens) an den Nachwuchs weitergegeben, z. B. beim Gen ALX1 beide das Allel B, so ist der Nachwuchs in Bezug auf das Gen ALX1 homozygot. Unterscheiden sich die Allele ist z. B. das mütterliche Allel B und das väterliche Allel P, so ist der Nachwuchs in Bezug auf das Gen ALX1 heterozygot.
Fast alle untersuchten Grundfinken in den Populationen mit stumpfem Schnabel (Großgrundfink und Opuntien-Grundfink auf der Insel Española) waren homozygot für das Allel B. Fast alle untersuchten Grundfinken in den Populationen mit spitzem Schnabel (Opuntien-Grundfink auf der Insel Genovesa und Spitzschnabel-Grundfink auf der Insel Wolf) waren homozygot für das Allel P. Die Allele B und P sind in diesen Grundfinkpopulationen jeweils fixierte Allele. Ein fixiertes Allel ist ein Allel, das die einzige Variante ist, die für dieses Gen in der gesamten Population existiert. Alle Tiere der Population sind für das fixierte Allel homozygot.
Besonders interessant ist, dass das Gen ALX1 beim Mittelgrundfink (Geospiza fortis) polymorph ist. Genetiker bezeichnen ein Gen als polymorph, wenn es für das Gen mehrere Allele innerhalb einer Population gibt. Feldbeobachtungen haben gezeigt, dass es bei dieser Art auch eine Vielfalt der Schnabelformen gibt. PP-homozygote Mittelgrundfinken neigten dazu, verhältnismäßig lange, spitze Schnäbel zu haben, BB-Homozygote hatten verhältnismäßig tiefe, stumpfe Schnäbel, während BP-Heterozygote mittlere Schnabelformen hatten.
„Das aufregendste und bedeutendste Ergebnis war, dass die genetische Variation des ALX1-Gens mit der Variation der Schnabelform nicht nur zwischen den Arten der Darwin-Finken, sondern auch zwischen den Individuen einer Art, dem Mittelgrundfink, verbunden ist”, erklärt Leif Andersson, der die Studie leitete.
Die ökologische Merkmalsverdrängung
Bei der ökologischen Merkmalsverdrängung geht es darum wie groß die Merkmalsunterschiede, wie z. B. der Unterschied in der Schnabelgröße, zwischen zwei ähnlichen Arten, in verschiedenen Lebensräumen sind. In einem Lebensraum, in dem eine Art allein vorkommt, ist sie der anderen Art äußerlich ziemlich ähnlich, da die Merkmalsunterschiede ziemlich klein sind. Die Arten können sogar sehr schwer voneinander zu unterscheiden sein. In einem Lebensraum, in dem die beiden Arten gemeinsam vorkommen, unterscheiden sich die beiden Arten äußerlich ziemlich stark, da die Merkmalsunterschiede groß sind. Die beiden Arten sind also im Überlappungsbereich leichter voneinander zu unterscheiden.
Um dieses Phänomen zu erklären, entwickelten die Evolutionsbiologen William Louis Brown Jr. und Edward Osborne Wilson 1956 die Theorie der ökologischen Merkmalsverdrängung [3]. Die Idee, die dieser Theorie zugrunde liegt, ist ganz einfach: Angenommen, zwei sehr ähnliche Arten kommen in Kontakt. Wenn die Ressourcen begrenzt sind, werden die Arten wahrscheinlich stark konkurrieren und ein mögliches Ergebnis ist der Wettbewerbsausschluss: Der besser angepasste Wettbewerber wird überleben und der schlechter angepasste aussterben. Aber eine alternative Möglichkeit ist, dass die natürliche Selektion in jeder Art diejenigen Individuen begünstigt, deren Merkmalsvariante, z. B. die Schnabelgröße, es ihnen erlaubt, Ressourcen zu nutzen, die nicht von Mitgliedern der anderen Art genutzt werden. Das Ergebnis kann sein, dass die Arten beginnen in Bezug auf Phänotyp und Ressourcennutzung zu divergieren (daher der Begriff Verdrängung), was den Ressourcenwettbewerb reduziert und eine Koexistenz ermöglicht.
Das Forscherteam von Leif Andersson hat so eine ökologische Merkmalsverdrängung bei Grundfinken auf der Galápagosinsel Daphne Major beobachtet und das damit verbundene Gen identifiziert [4]. Auf Daphne Major teilen sich der Mittelgrundfink (Geospiza fortis) und der Großgrundfink (Geospiza magnirostris) einen Lebensraum und ernähren sich von Samen. Zwischen den Jahren 2004 und 2005 kam es zu einer großen Dürre und das Angebot an großen Samen wurde knapp. Die Mittelgrundfinken mit großen Schnäbeln waren nun gegenüber den Großgrundfinken benachteiligt und starben aus. In der Folge nahm die durchschnittliche Schnabelgröße in der Mittelgrundfinkpopulation im Laufe der Generationen stark ab.
Die Genetiker verglichen die DNA-Sequenzen von Mittelgrundfinken, die die Dürre überlebt hatten, mit denen von Mittelgrundfinken, die während der Dürre gestorben waren. Sie fanden die meisten Unterschiede in dem Gen HMGA2 (high mobility AT-hook 2). Das Gen lag in zwei Allelen, L und S, vor. Mittelgrundfinken mit großem Schnabel waren homozygot für das Allel L (l=large). Mittelgrundfinken mit kleinem Schnabel waren homozygot für das Allel S (s=small).
Wie erwartet hatten anteilsmäßig mehr SS-Individuen (mit kleinen Schnäbeln) die Dürre überlebt während anteilsmäßig mehr LL-Individuen (mit großen Schnäbeln) gestorben waren. Die heterozygoten LS-Individuen zeigten anteilsmäßig eine mittlere Überlebensrate, was für einen additiven genetischen Effekt spricht.
HMGA2 ist ein Chromatin-assoziiertes Protein, dem die intrinsische Transkriptionsaktivität zu fehlen scheint, dass aber die Wirkung anderer Transkriptionsfaktoren verstärkt. „Das HMGA2-Protein modifiziert die Expression von Genen, aber der genaue Mechanismus, wie es die Schnabelgröße bei Darwinfinken kontrolliert, ist unbekannt”, so Leif Andersson.
Fußnote
1. HMS steht für “His Majesty’s Ship”. Die Reise führt über die Kapverdischen Inseln, entlang der Ost- und der Westküste Südamerikas, zu den Galápagosinseln, nach Tahiti, Neuseeland, Tasmanien, Mauritius, Kapstadt, nochmals Südamerika und über die Azoren zurück nach England.
2. Das Gen ALX1 ist auch ein Hox-Gen. Hox-Gene haben eine spezifische 180 Basenpaare lange DNA-Sequenz, eine sogenannte Homöobox, die für die Homöodomäne (60 Aminosäuren) eines Proteins codiert. Mit dieser Homöodomäne bindet das Protein an eine spezifische DNA-Sequenz und reguliert so die Genexpression. Die Hox-Gene sind im Genom gruppenweise angeordnet und codieren eine Klasse von Transkriptionsfaktoren deren Expression räumlich und zeitlich aufeinander abgestimmt ist. Die Reihenfolge der Hox-Gene auf dem Chromosom entspricht der Reihenfolge ihrer Expression entlang der Längsachse des Embryos. Gene am Anfang der Hox-Gruppe werden in frühen Entwicklungsstadien und weiter vorn im Embryo exprimiert, weiter hinten gelegene Gene dagegen werden in der Entwicklung später und in den hinteren Körperteilen aktiviert. Hox-Gene wurden bisher in allen untersuchten Tieren, sowie bei Pilzen und Pflanzen gefunden. Schwämme besitzen nur ein einziges Hox-Gen, bei Gliederfüßern sind es acht und beim Menschen sind es vier Hox-Gengruppen (Hoxa, Hoxb, Hoxc, Hoxd) mit insgesamt 39 Hox-Genen, die auf vier Chromosomen liegen; das sind die Chromosomen 2 (Hoxd), 7 (Hoxa), 12 (Hoxc), 17 (Hoxb). Evolutionsgenetiker nehmen an, dass Unterschiede in Kombination und Expression von Hox-Genen teilweise für die unterschiedlichen Körperbaupläne der Tierstämme verantwortlich sind. Spezialisierte Entwicklungsgene wie die Hox-Gene sind von den Wirkungen anderer Gene weitgehend unabhängig und ermöglichen die eigenständige Entwicklung bestimmter Strukturen bzw. Organe des Embryos. Das Hox-Gen Pax-6 ist z. B. für die Entwicklung des Auges bei Säugetieren nötig. Das erklärt, warum die sogenannte Mosaikevolution ein so verbreitetes Phänomen ist.
Weiterführende Literatur
Darwinfinken sind keine Finken: Makroevolution revisited


Polymorphe Allele wie die Gene ALX1 und HMGA2 sind also Schalter, die es einer biologischen Art erlauben in relativ kurzer Zeit einem Grossteil der Population eine andere Erscheinungsform zu geben, einfach indem Umweltfaktoren eines der Allele selektioniert. Im oben von Joe Dramiga erwähnten Beispiel mit dem Mittelgrundfinken, dessen Population in wenigen Jahre nach der Dürre 2005/2006 kleinschnabeliger wurde, wurde also eines der bei dieser Art vorkommenden polymorphen Allele begünstigt und die Erscheinungsform der Mittelgrundfinken änderte sich in kurzer Zeit.
Damit sind also schnelle Populationsanpassungen an geänderte Umweltbedingungen möglich. Denn dabei spielt Selektion die entscheidende Rolle und diese betrifft ja alle Nachkommen der aktuellen Population während der Weg über Mutation und dann Selektion viel langsamer ist, denn die Mutation betrifft meist nur gerade ein Individuum. Bis sich eine zufälligerweise nützliche Mutation über die ganze Population verbreitet hat, dauert es viel länger als bei der Selektion eines bereits in der Art vorkommenden polymorphen Allels.
Ich könnte mir vorstellen, dass bestimmte Arten sich über solche polymorphe Allele auch relativ schnell an eine Klimaänderung anpassen können. Die jetzige Erdsystemerwärmung beispielsweise wird einige Arten an bestimmten Orten aussterben lassen, andere Arten aber werden sich anpassen, wobei polymorphe Allele wohl eine Rolle spielen.
Übrigens tauchen die Darwinfinken auch im Wikipedia-Eintrag Polymorphism als Beispiel eines Polymorphismus auf. Ein anderes Beispiel dort ist der helle und der dunkle Jaguar.
Der 2013 veröffentlichte Artikel Evolution of a genetic polymorphism with climate change in a Mediterranean landscape. zeigt am Beispiel des echten Thymians (Thymis vulgaris), dass die kältesensible Variante dieser Gewürzpflanze seit 1970 in der Mittelmeerregion deutlich abgenommen hat. Klar: Warum den Aufwand für Gefrierschutz treiben, wenn es das doch kaum noch braucht, weil solch tiefe Temperaturen rund ums Mittelmeer kaum noch vorkommen.
Nach Richard Dawkins kann Evolution als ein Spiel der Allele aufgefasst werden, die mithilfe der von ihnen gebildeten Phänotypen um ihr Überleben “kämpfen”. Ein Allel ist dann erfolgreich, wenn sich das Individuum, in dessen Körper, sich das Allel befindet, erfolgreich fortpflanzt, was natürlich vom Phänotyp abhängt.
Darf ich mir erlauben, eine Frage zu stellen, die nicht direkt mit dem Artikel zusammenhängt?
Gibt es eine Regel, nach der Namen wie ALX1 oder BRCA1 vergeben werden? Kann man aus dem Namen in irgendeiner Form auf die Funktion schließen?
Die wichtigsten Regeln besagen,
dass der Name eines Gens aus drei bis fünf Zeichen bestehen soll
nur lateinische Buchstaben und/oder arabische Zahlen beinhaltet
mit einem Großbuchstaben beginnt, gefolgt von Kleinbuchstaben (bei Nagern)
in Publikationen kursiv geschrieben wird
Da es sich bei diesen Gennamen, um Abkürzungen handelt, kann man manchmal auf der biochemischen Ebene auf die Funktion schließen wie z. B, Lepr = Leptin Rezeptor. Welche Funktion der Leptinrezeptor im Fettstoffwechsel oder bei Fettleibigkeit hat ist damit nicht gesagt. Die Namen von Genmutationen sagen allerdings häufig etwas über ihre Auswirkung auf den Organismus und damit indirekt etwas über die Funktion des Gens auf höherer Ebene aus. Es gibt z. B. die Mutation Tinman (Blechmann), wenn ein Maus-Embryo diese Mutation hat, wird er sich ohne Herz entwickeln, genau wie der Blechmann aus dem Kinderbuch “Der Zauberer von Oz” von Lyman Frank Baum. Eine andere Mutation trägt den Namen Casanova. Zebrafische mit dieser Mutation werden mit zwei Herzen geboren. Es kommt vor, dass Forscher in der Natur eine Mutation entdecken ohne genau zu wissen welches Gen genau mutiert ist, sie wissen dann vielleicht nur auf welchem Chromosom sich die Mutation befindet.
Gibt es da ähnliche Befunde bei den auf den Hawaii-Inseln endemischen Kleidervögeln?
Ich habe bisher nicht von ähnlichen Befunden bei den Kleidervögeln auf den Hawaii-Inseln gehört. Es ist vermutlich auch schwieriger solche Befunde zu erheben: Die Kleidervögel haben zwar auch wie die Darwinfinken eine adaptive Radiation durchlaufen, aber von den 34 Arten sind bereits etwa die Hälfte ausgestorben; die übrigen sind entweder vom Aussterben bedroht oder stark gefährdet.