Darwinfinken sind keine Finken: Makroevolution revisited

Die ersten Naturforscher hatten keinerlei Vorstellung von der großen Formenvielfalt des Lebens auf der Erde. Sie kannten nur die Tiere und Pflanzen ihrer unmittelbaren Umgebung. Das änderte sich mit den Entdeckungsreisen, die sie vom 16. bis 19. Jahrhundert unternahmen: Es zeigte sich, dass jeder Kontinent seine eigene, einheimische Tier-und Pflanzenwelt besitzt und dass es auch je nach der geografischen Breite große Unterschiede in der Umwelt gibt: Die Natur sieht in den Tropen ganz anders aus als in den gemäßigten und arktischen Regionen. Um zu verstehen, wie neue Arten entstehen, begannen die systematisch orientierten Forscher verschiedene Populationen einer Spezies aus unterschiedlichen geografischen Regionen zu vergleichen. Das taten sie am gründlichsten bei Säugetieren und Vögeln und entdeckten dabei verschiedene Mechanismen der Artbildung.
Darwinfinken als Musterbeispiel der adaptiven Radiation
Wenn aus einer einzigen Stammart viele neue Arten entstehen, dann sprechen Evolutionsbiologen von Radiation. Wenn gleichzeitig mit dem Anstieg der Artenzahl auch die ökologische Vielfalt der Lebensräume zunimmt, in denen diese Arten leben, dann sprechen sie von adaptiver Radiation.
In allen Biologiebüchern der gymnasialen Oberstufe werden als Musterbeispiel für die adaptive Radiation die Darwinfinken genannt: Sie leben auf manchen Inseln des Galápagos-Archipels1 und bilden 18 eng verwandte Arten, die alle von einem gemeinsamen Vorfahren abstammen. Darwinfinken sind keine Finken2, wie der Name nahelegt, sondern gehören zu den Tangaren (Thraupidae): Das ist eine Familie der Singvögel, die zur Ordnung der Sperlingsvögel (Passeriformes) gehört. Die Gattung Grundfinken (Geospiza) sucht überwiegend am Boden Nahrung, die Gattung Baumfinken (Camarhynchus) in Bäumen und Sträuchern.
Die Schnabelformen der Darwinfinken
Darwinfinken sehen untereinander viel verschiedenartiger aus als die verwandten kontinentalen Tangaren gleichen evolutionären Alters. Darwinfinken unterscheiden sich in der Durchschnittsgröße ihres Schnabels und in der Schwankungsbreite seiner Abmessungen. Bewohnt eine Darwinfinkart allein und ohne Konkurrenz durch andere eine Insel, ist die Schwankungsbreite bei der Schnabelgröße viel größer und reicht dann bis in einen Bereich, der an anderen Orten von einer konkurrierenden Art besetzt wird3. Die Vogelkundler sahen auch, dass Arten, die sich in ihrer Nahrung unterscheiden auch in ihren Schnabelformen unterscheiden. Die samenfressenden Arten haben dicke, klobige Schnäbel, Mischnahrung- und insektenfressenden Arten schmalere, spitze Schnäbel. Das kann man sehr gut beim Großgrundfink (Geospiza magnirostris), einem Samenfresser und beim Waldsängerfink (Certhidea olivacea), einem Insektenfresser, sehen.
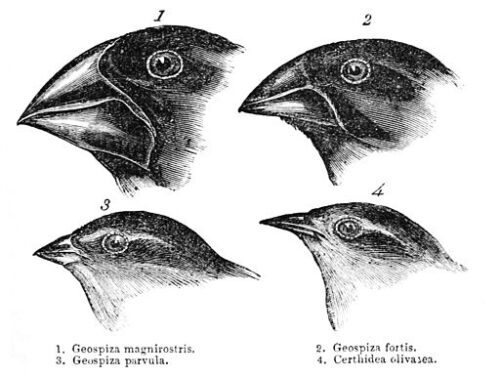
Die Evolutionsbiologen nahmen nun zwei Dinge an: erstens, dass die spezifische Schnabelform eine Anpassung an die spezifische Nahrung ist und zweitens, dass diese Form-Funktionsbeziehung für alle Vogelarten gilt. Doch diese Annahmen wurden nie überprüft.
Der Biologe Guillermo Navalón von der Universität Bristol in England, hat nun gemeinsam mit Kollegen in Spanien und den USA diese Annahmen überprüft und die Ergebnisse ihrer Studie in der Fachzeitschrift Evolution veröffentlicht [1]. In Schädeln von 176 Vogelarten untersuchten sie zum einen die Form des oberen Schnabels mit der geometrischen Morphometrie zum anderen die Hebelwirkung des zum oberen Schnabel ziehenden Kiefermuskels des unteren Schnabels. Diese Hebelcharakteristik spiegelt den Grad wider, in dem der Schnabel so angepasst ist, dass er starke Bisskräfte erzeugt. Anhand der Literatur zur Ernährung, Nahrungsbeschaffung und zum Fressverhalten untersuchten die Forscher, wie sich diese beiden Schnabeleigenschaften in Bezug auf die gesamte Ernährungsökologie entwickelten. Anschließen versuchten sie mit einer Phylogenetischen Vergleichsanalyse die Evolutionsgeschichte einer Schnabelform über die Abstammungsgeschichte der Vogelart herzuleiten. “Wir haben uns eine riesige Auswahl an Schnabelformen angesehen: Kolibris, Adler, Papageien, Papageientaucher, Flamingos, so ziemlich jeden Schnabel, den Sie sich vorstellen können.”, so Navalón, Erstautor der Studie.
Die Vogelkundler fanden einen signifikanten Zusammenhang zwischen der Schnabelform und der Ernährungsökologie bei modernen Vögeln. Die Variation in der Ernährungsökologie machte jedoch nur einen relativ kleinen Teil der Variation in der Schnabelform aus: Die Variation in der Verwendung des Schnabels beim Fressen erklärte beispielsweise nur etwa 9 % der gesamten Variation der Schnabelform, und die Variation in der Nahrung machte 17 % der gesamten Variation der Hebelwirkung aus. Darüber hinaus zeigten die Forscher, dass Arten mit unterschiedlicher Ernährung manchmal mit ähnlich geformten Schnäbeln ausgestattet waren.
Da die Ernährungsökologie die Vielfalt der Schnabelformen nur schlecht erklärt, vermuten die Forscher, dass funktionelle und ökologische Kosten-Nutzen-Abwägungen einen starken Einfluss auf die Schnabelevolution gehabt haben. Das ist zu erwarten, da der Schnabel neben dem Fressen einer Vielzahl Aufgaben dient, wie z. B. Singen, Putzen, Kämpfen, Trinken und Wärmeregulation. Da diese Aufgaben unterschiedliche (und manchmal widersprüchliche) Schnabelformen erfordern, können funktionale Kompromisse auftreten, die die Entwicklung der Schnabelform einschränken. Darüber hinaus kann die relative Bedeutung der einzelnen Funktionen je nach Vogelart stark variieren (z. B. singen einige Vögel selten), was zu unterschiedlichem Selektionsdruck zwischen den Arten führt.
„Der Zusammenhang zwischen Schnabelformen und Ernährungsökologie bei Vögeln war viel schwächer und komplexer als wir erwartet hatten. Diese Ergebnisse haben wichtige Auswirkungen auf die Untersuchung fossiler Vögel. Wir müssen vorsichtig sein, wenn es darum geht, die Ökologie bei ausgestorbenen Vögeln abzuleiten, die wir oft nur aufgrund der Form des Schnabels annehmen.“
Navalón
Zellen der Neuralleiste bestimmen die Schnabelform
Die Änderung der Schnabelform beruht auf Änderungen in bestimmten Zellgruppen der Neuralleiste, die sehr schnell erfolgen können. Die Zellen der Neuralleiste wandern während der Embryonalentwicklung in den vorderen Teil des Kopfes. Dort bilden sie fünf Zellbereiche, die mit der lokalen Oberhaut interagieren und zu den beiden schnabelförmigen Kiefern heranwachsen (wobei der obere Kiefer aus drei und der untere aus zwei der Zellbereiche entsteht). Transplantationsexperimente zeigen, dass die Schnabelform von der autonomen Aktivität der Neuralleistenzellen bestimmt ist: Wenn diese Zellen von einem Wachtelembryo auf einen Entenembryo übertragen werden, bildet diese Schimäre den Schnabel des Wachteltyps und umgekehrt. Der Selektionsdruck auf die Änderung der Schnabelform ist eigentlich ein Selektionsdruck auf die Änderung des Verhaltens der Neuralleistenzellen.
Fußnoten
1. Die Galápagosinseln liegen etwa 1000 km westlich der Küste Ecuadors und besitzen eine einzigartige Artenvielfalt. 1978 wurde die Inselgruppe von der Unesco zum Weltnaturerbe erklärt. Als Inseln vulkanischen Ursprungs können sich die Darwinfinken nicht auf den Inseln entwickelt haben, sondern müssen ihren Ursprung auf dem Festland haben. Sie haben sich vor 2,3 Millionen Jahren auf den Galápagosinseln angesiedelt. Aufgrund des Verwandtschaftsgrads zwischen den einzelnen Arten und der genetischen Homogenität können wir abschätzen, dass die Inseln ursprünglich von einem Schwarm aus etwa 30 Individuen besiedelt wurden.
2. Der Begriff „Darwinfinken“ wurde 1936 von Percy Roycroft Lowe (1870–1948) geprägt und durch das 1947 erschienene Buch Darwin’s Finches von David Lack populär gemacht.
3. Darüber hinaus ist das Größenspektrum der Samen, die sie verzehren, viel breiter als bei den Festlandtangaren: Genauer gesagt, finden wir auf ihrem Speiseplan alle auf den Inseln verfügbaren Größen von Samen. Möglicherweise nutzen Festlandtangaren nur einen kleinen Teil des Samenangebots, weil sie sich dieses mit anderen Vögeln und auch mit Nagetieren teilen müssen.


Die Kladistik ist halt ein “heißes Eisen”, Ihr Langzeit-Kommentatorenfreund, Joe, dankt für Ihre Nachricht, sog. Schnabel sind Instrumente, die nicht art-bestimmend sein können.
Mit freundlichen Grüßen und weiterhin viel Erfolg.
Dr. Webbaer
@ Joe Dramiga
Zitat: „Die Änderung der Schnabelform beruht auf Änderungen in bestimmten Zellgruppen der Neuralleiste, die sehr schnell erfolgen können. Die Zellen der Neuralleiste wandern während der Embryonalentwicklung in den vorderen Teil des Kopfes. Dort ….“
Es scheint, dass der zitierte Mechanismus sehr grundlegend im Zusammenhang mit der Genexpression ist.
Ich würde Sie gerne um Antwort zu den nachstehenden Fragen bitten:
Gibt es hauptsächlich zusammenhängende Bereiche auf der „linearen Stammzellen DNA“, die entweder von einem immer gleichartigen (oder eventuell auch verschiedenen) chemischen Funktionsmuster gesteuert, die jeweiligen DNA Bereiche aktivieren, so dass die jeweiligen Zellverbände, z.B. der Neuralleiste, gebildet werden und auch die Anpassungsprozesse an die benachbarten Zellverbände gesteuert werden?
Es ist demnach die Frage ob es sich ähnlich verhält wie bei einem „Unterprogramm“ (oder einer „Funktion“ oder einem „Prozess“ wie in der Informatik. Durch oftmalige Aktivierung eines bestimmten “Programmteiles” (“Unterprogrammkonzept”), kann man “Programmcode “einsparen”, man erhält wesentlich “kürzere Programme”.
Die Stammzellen DNA würde einem „Hauptprogramm“ der Informatik entsprechen, die jeweilige Funktion (Unterprogramm) einen Teil des Hauptprogramms, dass oftmals „aufgerufen“ (aktiviert) wird um eine Vielzahl immer gleicher Zellen bzw. Zellverbände zu erzeugen.
Wäre es so, würde dieses „Unterprogrammkonzept“ erklären, warum z.B. die menschliche DNA „kürzer“ ist als die DNA z.B. einer Banane, was irgendwie absurd erscheint.
@Elektroniker Ich verstehe deine Fragen nicht ganz, aber ich versuche so gut es geht zu antworten.
*gleichartigen (oder eventuell auch verschiedenen) chemischen Funktionsmuster*
In der befruchteten Eizelle wird z. B. die Kopf-Schwanz-Achse durch sogenannte Morphogene festgelegt. Morphogene sind regulatorische Proteine im Zytoplasma der Eizelle, die jeweils zu den gegenüberliegenden Polen der Eizelle diffundieren und so Konzentrationsgefälle entlang der Kopf-Schwanz-Achse bilden (und natürlich auch entlang der anderen beiden Raumachsen). Wenn sich die Eizelle teilt, dann haben die Tochterzellen eine unterschiedliche räumliche Verteilung der Konzentrationen der Morphogene in ihren Zytoplasmen. Dadurch erhalten die Tochterzellen Informationen über ihre Position im Embryo.
*zusammenhängende Bereiche auf der „linearen Stammzellen DNA“*
Die Morphogene in den Tochterzellen wiederum aktivieren bestimmte Regulationsgene in den Zellen. Ein Beispiel für solche Regulationsgene sind die Hox-Gene. Sie bestimmen die Grundeinteilung des Körpers in Kopf und Rumpf. Die Hox-Gene sind auf dem Chromosom dicht hintereinander angeordnet. Die Anordnung der Hox-Gene auf einem Chromosom entspricht der Reihenfolge der Körperzonen, für die sie “zuständig” sind. Das Hox-Gen was für den vorderen Bereich des Körpers zuständig ist liegt vorne auf dem Chromosom, das was für den hinteren Bereich des Körpers zuständig ist liegt hinten auf dem Chromosom.
@ Joe Dramiga
Danke für Deine Ausführungen, sie haben mir sehr beim Verständnis der Vorgänge geholfen.
Wie die Tochterzellen die Informationen über ihre Position im Embryo erhalten ist recht einleuchtend erklärt.
Es gibt aber noch die Frage nach dem zeitlichen Aspekt. Man kann zwar davon ausgehen, dass ein neuer Prozess eben dann beginnt wenn der vorherige Prozess einen bestimmten geeigneten Status erreicht hat, aber es könnten auch zeitliche Aspekte eine Rolle spielen. Wenn z.B. eine Pause eingelegt werden muss, damit sich gerade laufende Prozesse voll entfalten können. (Anschaulich: Wenn z. B. der Beton trocken sein muss ehe weiter gebaut wird, oder beim Bäcker der Teig „rasten“ muss bis er weiter verarbeitet werden kann).
Und da hätte ich die Vermutung, dass die nicht codierende DNA beim jeweiligen Expressionsprozess eine gewisse Zeit benötigt und wenn diese Zeitspannen „abgelaufen“ sind, wieder „normale Eiweiße“ erzeugt werden, die sozusagen „an der Reihe“ sind.
Individuelle Muster in den körperlichen Ausprägungen, z.B. von den Fingerabdrücken bis zum jeweiligen Gesichtsausdruck der verschiedenen Menschen könnte so zustande kommen.
Gibt es darüber in der Wissenschaft Erkenntnisse?
Die individuelle Merkmalsausprägung hängt nicht nur von den Erbanlagen ab, sondern wird auch noch durch Umwelteinflüsse und physiologische Einflüsse geprägt.
Mir geht es um die grundsätzliche zeitliche Struktur der bei der Genexpression auftretenden Prozesse.
Ich habe mich beruflich vor Jahrzehnten mit der elektronischen Steuerung technischer Prozesse beschäftigt. Es gibt gewisse systemische Ähnlichkeiten mit den Abläufen biologischer Prozesse. Allerdings liest man so gut wie nichts über die zeitliche Struktur dieser Prozesse in der Biologie. In der Technik ist die Zeitstruktur, neben anderen Mechanismen, von sehr großer Bedeutung.
Es ist für mich denkunmöglich dass die Zeitstruktur bei genetischen Prozessen keine Rolle spielt.
Besonders vermute ich, dass jeweils wenn die genetische Information der einzelnen Codons nacheinander abgelesen und umgesetzt wird, Zeit „verbraucht“ wird. Auch bei der Ablesung der nicht codierenden DNA. Wenn es nur um die „zeitliche“ Steuerung geht, dürfte es weiters auch unerwünscht sein, wenn Eiweiße entstehen könnten.
Im weitesten Sinne dürfte die zeitliche Steuerung auch zusätzlich (neben bekannten anderen Möglichkeiten) dafür sorgen, dass nicht einfach funktionslose riesige „Zellhaufen“ entstehen, sondern letztlich bestens strukturierte lebensfähige biologische Systeme.
Natürlich ist für mich nahe liegend, dass Firmen die höchst erfolgreich auf diesem Gebiet arbeiten über ihre Forschung nichts verlauten lassen und ihre Mitarbeiter unter strengster Geheimhaltung stehen.
Andererseits könnte dieser zeitliche Steuerungsmechanismus, besonders auch im Zusammenwirken mit Genmutationen, von Bedeutung sein.
Wenn z.B. in einer mehr oder weniger langen zeitlichen „Steuerkette“ von nicht codierender DNA ein für Mutationen „anfälliges“ Gen plötzlich zu einem Eiweiße codierenden Gen wird und unnötige, womöglich sehr schädliche Eiweiße, letztlich Zellgruppen generiert werden, so kann das ernsthafte Folgen haben. Es hängt aber noch von der „Geschwindigkeit“ der Prozesse ab, die wiederum von der jeweiligen benötigten „Zeitspanne“ die bei der Expression der jeweils beteiligten nicht codierenden DNA abhängig ist. Der unerwünschte Expressionsprozess (letztlich der Krankheitsverlauf) kann demnach sehr langsam oder auch sehr schnell sein.
In diesem Sinne scheint es mir zweckmäßig, die „Zeitstruktur“ der biologischen Prozesse (Genexpression) auch in der Biologie zu erforschen.
Natürlich wird in der Biologie und Medizin auch der zeitliche Aspekt der Genexpression untersucht – nur ist in lebenden Vielzellern normalerweise die Genexpression schwierig in real time zu erfassen, weil sie schwierig zu beobachten ist. Eine zeitliche Auflösung im Minutenbereich kann ich mir allerdings in speziellen Fällen und mit speziellen Methoden vorstellen.
Lies dazu mal meinen Blogartikel Genexpression im Gehirn live beobachten! da siehst du was für ein technischer Aufwand betrieben werden muss. Generell ist es bei Proteinen, die im Blutkreislauf zirkulieren UND eine lange Halbwertszeit haben, einfacher so etwas zu messen als z. B. bei Proteinen, die sich im Gehirn befinden und kurzlebig sind. Wieviel Zeit vergeht zwischen dem Verzehr einer Bratwurst und der Expression des Insulingens in den Beta-Zellen des Pankreas und wie lange wird diese Expression aufrechterhalten? Niemand weiß es. Bessere Chancen, was die zeitliche Auflösung betrifft, haben Forscher da in der Zellkultur z. B. mit lichtregulierter Genexpression bei lichtempfindlichen Zellen.
Es ist sicherlich ein großes Problem der Genforschung dass die Prozesse der „Genexpression“ derzeit nur schwierig in „real time“ zu erfassen sind.
Es ist der ganz große Vorteil der Elektronik, dass sie die elektronisch gesteuerten, derzeit hauptsächlich technischen Prozesse, bestens „im Griff“ hat. Besonders der zeitliche Aspekt ist bei den elektronisch gesteuerten technischen Prozessen besonders gut (z.B. mittels Oszilloskopen) zu beobachten. Technische Prozesse für sehr viele Fachgebiete können bestens gesteuert, überwacht und auch analysiert werden.
Allerdings frage ich mich, ob man besonders auf „elementarer Ebene“, was z.B. die „Aufgaben“ der nicht codierenden DNA betrifft, derzeit noch zu wenig weiß?
Einerseits dürfte auch derartige DNA z.B. bei der Transkription (zu „Lasten“ aber auch zum besseren “Gelingen“ der nachfolgenden Prozesse) „Zeit“ benötigen, andererseits dürfte auch bei der nicht codierenden DNA, RNA entstehen, die ebenfalls Einfluss auf das Geschehen nehmen könnte, auch wenn zumindest direkt, keine Eiweiße entstehen.
Man hat probeweise aus DNA vermeintliche „Junk DNA“ (früher ging man davon aus, nicht codierenden DNA wäre immer „Junk“ (Müll)) entfernt, und es waren keine Folgen zu erkennen.
Daraus könnte man Schlüsse ziehen, es ist tatsächlich auch echte „Junk DNA“ vorhanden, oder Auswirkungen könnte es geben, können aber nicht erkannt werden, können jedoch trotzdem vorhanden sein. Allenfalls wurde „heutige Junk“, also nicht (mehr) codierende DNA, früher (z.B. für die Systementwicklung) gebraucht. (Wie z.B. Bei einem Automotor mitunter besondere „Haken“ vorhanden sind um den Motor mit einem Kran ein- bzw. ausbauen zu können, die aber für die Funktion als Motor nicht erforderlich sind).
Will damit sagen, besonders Biotech Firmen die mit der Erzeugung z.B. künstlicher Haut (Brandverletzungen), oder künstlichem Fleisch beschäftigt sind, sollten eigentlich z.B. die zeitlichen Auswirkungen der „Junk DNA“ erforschen können, indem sie einerseits nachforschen ob Prozesse bei bestimmten „herausgeschnittenen“ nicht codierenden DNA Bereichen schneller verlaufen, eventuell auch Probleme bei der Zellentstehung auftreten, oder andererseits ganz egal welche nicht codierenden Abschnitte künstlich entfernt oder in das Prozessgeschehen eingebunden werden, sich am Ergebnis der Prozesse überhaupt nichts ändert?
Es geht darum verschiedene Varianten von nicht codierender DNA korrekt (allenfalls nach einem „Start Codon“ und vor einem „Stopp Codon“) in die relevanten Prozessketten einzuschleusen, bzw. vorhandene nicht codierende DNA zu entfernen, um aus dem veränderten zeitlichen Verhalten der Prozesse oder an den veränderten strukturellen Eigenschaften der entstehenden Zellverbände, Aussagen zum Prozess steuernden Verhalten von Junk DNA machen zu können.
Wären eindeutige Aussagen möglich, könnte man früher oder später genauso gut chemische Prozesse mit einem „DNA Programm“ planmäßig steuern, wie heutzutage elektronisch Prozesse aus nahezu allen Bereichen der Wissenschaft (besonders auch Technik und Wirtschaft) perfekt von „Software“ gesteuert werden können.
Wie ehemals Informatiker leistungsfähige und komfortable „Programmierwerkzeuge“ und Programmbibliotheken zur kostengünstigen Massenproduktion von Software entwickelt haben, so könnten auf das vorhandene Wissen aufbauend, Konzepte und Methoden zur präzisen Steuerung biologischer Prozesse entwickelt werden.
Nach der Entwicklung der „Genschere“ (für das „Genom Editing“) kommt es darauf an, die systematische Prozesssteuerung bei genetischen Prozessen vorerst grundlegend, später auch im Detail zu verstehen und vor allem auch, wegen der extremen Komplexität, im Computer „abbilden“ und „simulieren“ zu können.
Dies wäre eine Grundlage für künftige Erfolge in der Biotechnologie.
Eine Frage. Warum heißt der Artikel “Makroevolution revisited”, wenn es doch um Variation geht und nicht um Innovation?
Der Titel wurde gewählt, weil es um Aussagen ging, die eine ganze Klasse von Wirbeltieren betrifft.