Eine neue Domäne?
BLOG: Bierologie

Das gesamte bekannte Leben wird in drei Domänen aufgeteilt – Bacteria, Archaea und Eukaryoten. Bacteria und Archaea kann man in Prokaryoten, also Lebewesen ohne Zellkern, einteilen, währen Eukaryoten über einen Zellkern verfügen (da sind wir Menschen auch drin!).
Archaea und Bacteria unterscheiden sich stark in der Struktur ihrer ribosomalen RNA voneinander und wurden deshalb in verschiedene Domänen unterteilt.Alle drei Domänen stammen von je einem Organismus ab.
Viren haben keine Domäne da ihr Status als Lebewesen umstritten ist – sie haben keine eigenen Ribosome, keinen eigenen Stoffwechsel und können sich nicht ohne Hilfe von Wirtsorganismen fortpflanzen.
Wir hatten hier vor geraumer Zeit einen Artikel über Mama- und Mimivirus – beides Viren, die außerordentlich groß sind, und über verschiedene Proteine verfügen, die vorher nur in zellulären Organismen bekannt waren. Gestern erschien bei PLoS one ein sehr interessantes Paper über die Gruppierung, in der u.a. Mimi- und Mamavirus eingeteilt sind, genannt NCLDV = nucleocytoplasmic Large DNA Viruses. NCLDV beinhaltet fünf Familien, die bis jetzt mit ähnlichen Eigenschaften bekannt sind. In der Veröffentlichung wird die Hypothese aufgestellt, dass NCLDV eine weitere Domäne darstellen könnte!
Dazu wurden verschiedene Proteome aus den drei Domänen ausgewählt und mit den bis jetzt veröffentlichten NCLDV-Proteomen verglichen, um so einen Stammbaum zu erzeugen:
Wens interessiert, hier ist die Abfolge der verwendeten Bioinformatik-Tools: BLASTP wurde verwendet, um homologe Proteine in den NCLDV-Proteomen zu finden; T-Coffee oder Muscle wurden für die multiplen Alignments verwendet (warum nich gleich nur Muscle ist mir noch schleierhaft); Gblocks wurde verwendet, um konservierte Blöcke zu finden (noch nie von mir benutzt). Maximum Likelihood Bäume wurden mit FastTree (JTT+CAT) berechnet, Bayessche Bäume wurden mit MrBayes (WAG + MCMC) erzeugt.
Hier der Baum für das Protein RNA Polymerase II, Beta-Untereinheit:
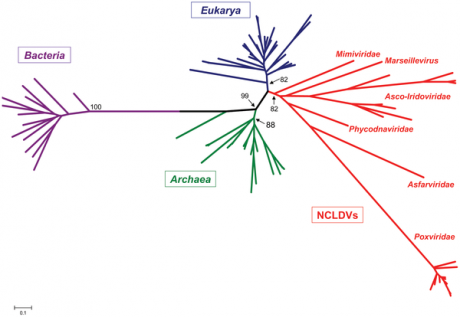
Quelle
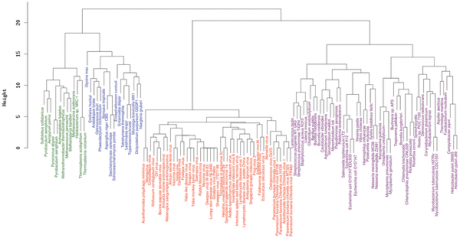
Hier sieht man bereits grob, dass sich die untersuchten Proteine in vier Gruppen unterteilen lassen, aber ab zum nächsten Bild: einem Baum, basierend auf Genen, die für Informationsunterbringung und – verarbeitung, sowie Nukleotidtransport und -metabolismus zuständig sind. Der Baum basiert auf "phyletic patterns", also nicht darauf, ob Gene von verschiedenen Spezien auf der Nukleotidebene ähnlich oder nicht sind, sondern auf der bloßen An- oder Abwesenheit (im Sinne von 1 oder 0).
Eukaryoten in blau, Bacteria in violett, Archea in grün und NCLDV in rot.
Das sind die "schönen" Bäume, also die, die ein Selbstständigmachen der NCLDV in der frühen Erdgeschichte unterstützen. Wer die restlichen Bäume sehen möchte, findet diese in "Supporting Information", leider sind diese nicht sehr aufschlussreich, was wahrscheinlich an den ausgewählten Proteinen liegt.
Gibt es also eine vierte Domäne? Noch schwer zu sagen, ich denke aber schon; mehr (vor allem gen-basierte) Forschung ist nötig, um das Bild der Domänen weiter abzurunden. Dafür fehlen im Moment leider die Daten – aus den vorhandenen Bäumen hätte man noch einen Supertree erstellen können (wie z.B. hier für Dinosaurier), einfach nur um zu schauen, ob sich ein Supertree in diesem Fall bereits lohnt oder ob mehr Daten nötig sind. Die Auswahl der Proteine schlägt hier auch startk zu Buche; die gleiche Studie mit anderen, aber trotzdem homologen Proteinen könnte leicht zu anderen Bäumen führen.


Eine neue Domäne…irgendwann ja!
Die Modifikation der Domänen wird irgendwann nötig sein. Nicht nur durch die Funde, die du hier erklärst, sondern auch durch ein Bakterium, über das ein Paper anfang des Jahres erschien: Es geht dabei um das Archaeenbakteriun Ignicoccus hospitalis, dass eine äußere und eine innere Membran besitzt, was normalerweise nicht charakteristisch für Einzeller ist. Eine zweite innere Membran würde darauf vermuten lassen, dass das Ablesen von Genen und der Energiestoffwechsel getrennt sind, womit es irgendwo zwischen Pro- und Eukaryoten eingeordnet werden müsste. Dieser Organismus könnte in der Evolution nun genau zwischen der Entstehung von Einzellern und Vielzellern eingeordnet werden und bestärkt die Theorie, dass wir aus Einzellern evolviert sind. Der evolutionäre Stammbaum vervollständigt sich also, was verdammt cool ist!
Interessant! Extremophile sind meines Wissens nach immer sehr “erfinderisch” – vielleicht ist die zweite Membran erst später entstanden, so dass I. hospitalis keine eigene Domäne wäre, sondern nur ein Sonderweg innerhalb der Archeae wäre. Dafür müsste man nur die Definition der Archeae ein wenig ändern und nur die Basierung auf 16S rRNA-Sequenz nehmen.
Vielleicht kommt die Doppelmembran auch von der Assoziierung mit Nanoarchaeum equitans (auch sehr interessant!) in irgendeinem Wege den mein Sonntags-10Uhr-Morgens-Gehirn einfach nicht auf die Reihe kriegt.