Exakte Wissenschaft in biomedizinischen Datenbanken
BLOG: Graue Substanz

Wer interdisziplinär publiziert, riskiert, nicht in biomedizinischen Datenbanken gelistet zu werden.
 Ein wissenschaftlicher Artikel mit Bezug zur Medizin ist nahezu wertlos, wenn er nicht in PubMed erscheint, denn ihn findet niemand. PubMed ist die größte medizinische Datenbank mit bisher fast 22 Millionen Artikeln aus dem gesamten Bereich der Biomedizin.
Ein wissenschaftlicher Artikel mit Bezug zur Medizin ist nahezu wertlos, wenn er nicht in PubMed erscheint, denn ihn findet niemand. PubMed ist die größte medizinische Datenbank mit bisher fast 22 Millionen Artikeln aus dem gesamten Bereich der Biomedizin.
Als ich beim Wissenschaftsverlag Elsevier nachfragte, warum ein bestimmter Artikel von mir nicht in PubMed gelistet wurde und wie ich in Zukunft sicher stellen kann, dass meine Artikel dort erscheinen, bekam ich zur Antwort, dass die Zeitschrift (“Physica D“), in der mein Artikel erschien, exakte Wissenschaft veröffentlicht und die Artikel dort nicht generell in PubMed aufgenommen werden sondern nur Beiträge mit “dramatischen biomedizinischen Auswirkungen”.
Dear Dr Dahlem,
Thank you for your e-mail.
As per advised by our colleagues, this journal (Physica D: Nonlinear Phenomena) is not a PubMed journal.
PubMed covers only the biomedical sciences, and does not include all journals in that field. The journal is a physics journal. It would not be in PubMed generally and an individual article would only appear in PubMed if it had dramatic biomedical implications.
They add that unfortunately free databases parallel to PubMed do not generally exist for the hard sciences.
Die Antwort ist schlicht Blödsinn. Wie soll man die “dramatic biomedical implications” verstehen? Arbeiten mit Nobelpreis verdächtigen Auswirkungen nehme ich an? Und wer nochmal genau entscheidet das zum Zeitpunkt der Veröffentlichung? Immerhin, bisher haben es sieben Artikel aus der Zeitschrift Physica D geschafft.
Gab es vielleicht sogar nur diese sieben Artikel, die eindeutig in den Bereich der Biomedizin fielen, unabhängig von ihrer Bedeutung? Auch das ist klar nicht der Fall, ich kenne bei weitem mehr. Sieben Artikel sind also nicht wirklich viel für eine renommierte Zeitschrift, die seit 1980 alle zwei Wochen erscheint und zwar mit dem Themenschwerpunkt “Nichtlineare Phänomene”, der viele biologisch relevante Themen abdeckt. Bei meinem eigenen Artikel zum Beispiel hätte man diesen Bezug auch nicht anzweifeln oder gar übersehen können (s.u.), es sei denn, ein automatisiertes System trifft die Entscheidung auf einer anderen Basis.
Ganz sicher bin ich mir zwar immer noch nicht. Ich denke aber, die Auflösung ist recht banal.
Wer beim Einreichen seines Manuskripts in der Physik versäumt, die richtigen PACS anzugeben, der kommt nicht in PubMed. Vermute ich zumidest. PACS steht für Physics and Astronomy Classification Scheme, eine Klassifikation für die Bereiche Physik und Astronomie, vom American Institute of Physics (AIP) herausgegeben. Die Codes 87.19.L- (für Neuroscience) und 87.19.X- (für Disease) hätten den Trick wohl gemacht. Wäre nur schön gewesen, dies auch vom Customer Support von Elsevier zu erfahren, oder, falls es doch anders läuft, wer auf welcher Basis diese Entscheidung trifft.
Leider sind in der Tat nur 14 meiner Veröffentlichungen in PubMed gelistet. Es scheint mir daher ein generelles Probelm zu sein. Denn bei mindestens elf weiteren wäre ein Erscheinen in PubMed gerechtfertigt gewesen, auch wenn diese Arbeiten in physikalischen Zeitschriften erschienen.
Das ist ein Problem. Ich selbst nutze sehr häufig PubMed für die Literatursuche. Andere machen es wahrscheinlich ebenso, ich kenne Mediziner, die ausschließlich PubMed nutzen und wir übersehen so manchmal wichtige Beiträge.
Es stellt sich vielleicht noch die Frage, ob nur Artikel übersehen werden, die zwar einen klaren Bezug zur Biomedizin haben mögen aber dennoch zu mathematisch sind, wie zumindest implizit in der Antwort angedeutet. Zum einem ist dies in vielen Fällen nicht so. Der besagte Artikel ist nur in Teilen mathematisch geprägt. Ich glaube auch generell nicht, dass man diesbezüglich eine Trennlinie anhand des Begriffs “exakte Wissenschaft” (“hard sciences”) ziehen kann.
Als Beispiel hier der besagte Artikel von mir und meinen Mitautoren.

(M. A. Dahlem, R. Graf, A. J. Strong, J. P. Dreier, Y. A. Dahlem, M. Sieber, W. Hanke, K. Podoll, and E. Schöll: Two-dimensional wave patterns of spreading depolarization: retracting, re-entrant, and stationary waves, Physica D 239,889 (2010). DOI)
Die Adressenliste zeigt allein den klaren Bezug zur Medizin, von der einen Physik-Adresse abgesehen: zwei Kliniken für Neurologie, ein Leibniz-Institut für Neurobiologie, ein Max-Planck-Institut für Neurologie und zwei Kliniken für Psychiatrie. Für den Fall, dass man den Bezug nicht anhand der Adressen festmachen will, zeige ich noch eine Abbildung*.
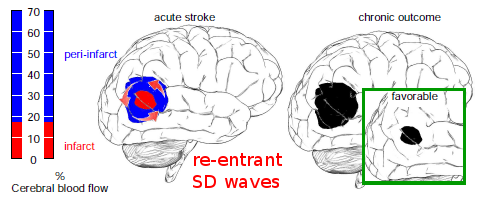
Es ist leicht zu erkennen, dass es um keine reine Physik handelt, nur für den Verlag leider nicht. Und nicht für potentielle Leser, die über PubMed nach Literatur suchen. Sie finden den Artikel nämlich erst gar nicht. Ob der Artikel nachträglich dort gelistet werden kann, weiß ich leider immer noch nicht. Den Customer Support von Elsevier brauche ich wohl nicht mehr zu fragen. Auch eine erneute Anfrage ergab schlicht die gleiche Antwort.
Fußnote
* Die Abbildung ist leicht verändert.


@Dahlem
wärs nicht einfach mal Zeit das papersystem abzuschaffen? ich behaupte, dass es immer mehr an die grenzen seiner komplexitätsklasse stößt und man versucht durch spezialisierung die grenze noch etwas rauszudrücken.
eine skizze für eine alternative hätte ich in der schublade. nötige werkzeuge sind schon vorhanden, man könnte morgenfrüh anfangen.
@Anton
“em>eine skizze für eine alternative hätte ich in der schublade“
Lass hören! Ich kenne auch kaum noch jemanden, der in der Bibliothek am Kopierer steht und Artikel aus Zeitschriften ausdruckt. Als Diplomand habe ich Stunden in Bibliotheken mit der Suche und Kopieren verbracht.
something wrong in the System
Ja, es läuft hier wohl was falsch im Staate der “World-science-community”. Was also “interdisziplinär” notwendig und Zielführend sei, ist noch nicht in alle Bereiche der Öffentlichkeitsarbeit durchgekommen. Dummerweise orientieren sich daran trotz besseren Wissens alle Beteiligten und richten ihr Paradigma danach aus. Was sollen sie auch sonst tun?
Da kann man wohl nichts machen – ausser die Schublade zu öffnen und in Vorleistung zu gehen.
Allerdings bin ich auch gespannt, was denn da in der Schublade als Lösung drinnen sei. Also bitteschön …. öffnen und vorlesen.
@Dahlem
Ehrlich gesagt fällt es mir schwer das in ein Kommentar runterzubrechen, ich könnte ohne probleme darüber 10 seiten schreiben, deswegen beschreibe ich es nur ganz kurz:
statt content in baumstrukturen (bspw. kapitel, unterkapitel) abzubilden, wird content auf vollständige gerichtete graphen abgebildet. die gedanklichen schritte zwischen knoten sollten möglichst klein sein. in der mathematik könnte das bspw. ein lemma-graph sein. es lassen sich dann weitere graphen parallel dazu bilden. bspw. ein hypergraph, der die lemma zu theorem zusammenfasst und noch ein weiterer paralleler graph, der übungen abbildet. dazu eignet sich org-mode aus dem sich direkt in latex/html etc. exportieren lässt.
dabei wird immer an einem einzigen projekt gearbeitet, dass mit hilfe von git gemanaged wird.
diese zwei sachen haben so viele implikationen, die ich garnicht hier alle aufzählen kann. insbesondere kennen zwar viele git, aber nicht die implikationen des konzepts dahinter. sodass die meisten leute keinen unterschied zwischen git und svn/cvs etc sehen….
das schöne an der sache ist, dass die beiden werkzeuge in der lage sind paper, textbooks(introductory und advanced) und übungen zu vereinen. weiter wird der arbeitsaufwand im vergleich nur für das papersystem massiv eingegrenzt und man könnte das projekt für seine eigene arbeit unmittelbar starten. man liest paper und erstellt dann direkt den graph als eine art exzerpt.
Der zweite Schritt
Sobald es nur noch elektronische Zeitschriften gibt, stellt sich natürlich die Frage, warum überhaupt noch in Heftstruktur denken oder, der zweite Schritt, in Artikeln.
Aber hier spreche ich doch etwas anderes an. Wer interdisziplinär publiziert (in welcher Form auch immer in Zukunft), riskiert, nicht in biomedizinischen Datenbanken gelistet zu werden, weil diese ja selektiv sein müssen. Klar erscheint in biomedizinischen Datenbanken nicht, zum Beispiel, ein Theorem zur Stabilisierung periodischer Orbits, obwohl dieses Theorem ja reichliche biomedizinische Anwendungen haben könnte.
Sollte nun solch eine Anwendung aber mit dem klaren Bezug zur Medizin publiziert werden, jedoch weiter in einem Physikheft, läuft man Gefahr, dass dies nicht wahrgenommen wird.
Publiziert man dies in einem Medizinheft, wird man von der anderen Community nicht für voll genommen.
Auch wenn Hefte vielleicht irgendwann verschwinden (glaube ich eigentlich nicht, sehe aber sofort ein, dass es gute Gründe dafür gäbe), Datenbanken werden immer dieses Problem haben.
@Dahlem
Wenn Sie einen Graphen einführen, haben Sie keinen Bedarf mehr Kategorien im Sinne fachlicher Grenzen einzuführen. Sie hätten einfach zwischen Physik und Medizin kaum Kanten. Bei ihrem speziellen Problem würde einfach ein Teilgraph zwischen Medizin und Physik liegen.
Wissenschaftler arbeiten dann effektiv nicht an kategorialen Artikel sondern auf Teilgraphen
@Anton
Wenn ich einen Graphen einführe. Da liegt das Problem. Wer macht das. Wer verwaltet diese Datenbank? PubMed wird von der United States National Library of Medicine verwaltet.
Wenn ich Dich hier richtig verstehe, sollte es dann eine allumfassende Datenbank geben. Aber selbst bei dieser muss jemand Artikel für Artikel oder Knoten für Knoten festlegen, welche Kategorien (Kanten) er hat.
Mein Artikel hätte da schlicht nicht per Default alle passenden bekommen. Oder?
@Dahlem
Die Verwaltung läuft über git. darin wird die komplette geschichte des graphen gespeichert und sämtliche forks lassen sich darin abspeichern. wenn sie bspw. zwei widersprechende modelle getrennt betrachten wollen, bspw. stringtheorie und standardmodel ist es mit git ohne probleme notwendig ein fork vom standardmodel zu machen und daraus einen stringtheorie-graphen zu machen. git hat unglaubliche fähigkeiten. bspw. können sie dann aktualisierungen im standardmodel direkt in die stringtheorie einfügen. das funktioniert fast vollautomatisch….
der clou an der idee ist dass das konzept selber extrem skalierbar ist. sie können es nur für sich selber benutzen. wenn sie ein paper lesen machen sie sich notizen dazu in dieser graphenform und können dann direkt aus org-mode in latex exportieren. das heißt sie würden mit relativ wenig aufwand aus ihren persönlichen notizen ein paper mit wenig aufwand schreiben können. gleichzeitig können sie aber dank git und graphenform den gesamten content der wissenschaft aufnehmen, selbst von sich widersprechenden theorien ohne diese konflikte explezit abzubilden…
ich kann da nicht weiter drauf eingehen…
@Dahlem
sry für die ganzen fehler in der antwort, mir fehlt halt die motivation das ordentlich zu machen, weil der impact ja sehr gering ist.
allein für git müsste man mehrere seiten schreiben, obwohl es relativ einfach funktioniert. ich rate ihnen diesen vortrag hier zu gucken:
http://www.youtube.com/watch?v=4XpnKHJAok8
auch wenn sie git niemals in ihrem leben benutzen werden sind die einsichten mehr als nützlich.
@Markus
Um nochmal auf ihre eigentliche Frage einzugehen: Mein Eingangsvorschlag war ja das papersystem abzuschaffen. Das kann ich natürlich nicht so schnell. Egal wie gut eine Idee ist, sie muss am Ende auch benutzt werden. Ihr konkretes Problem liesse sich lösen, wenn genügend Wissenschaftler solche Graphen lesen würden und der Weg dahin ist, dass Sie diese arbeitsweise erstmal nur für sich benutzen. Dann geben Sie ihn weiter an weiter an Kollegen und Studenten, die sich einarbeiten müssen etc… Aus dem Graph erzeugen Sie Paper und verlinken die Paper auf ihren Graph. Falls einer ihrer Studenten Änderungen vornimmt können Sie diese unter vollautomatisch und voller Kontrolle in ihren Graphen reimportieren. So setzt sich langsam ein Schneeballsystem in Gang. Sie führen eine zweite “Ebene” ein. Ein Graph, der Übungen abbildet für ihre fortgeschrittenen Vorlesungen und bieten einen Fork ohne Lösungen(sonst komplett) als Vorlesungsmaterial an. Irgendwann haben Sie genug paper verarbeitet, dass Sie eine Art fortgeschrittenen “Buch” für ihr Spezialgebiet haben und das wird mehr Leute motivieren ihren eigenen Graph zu übernehmen und zu mergen. Am Ende des Tages entstehen dann ähnliche Strukturen wie beim Linuxkerneldevelopment.
git
Ich kenne git. (Auch wenn ich bisher svn nutze.)
Sobald man von der Artikelstruktur weg ist, muss man natürlich auch die Zahl der bisher 22 Millionen Artikeln aus dem Bereich der Biomedizin hinterfragen. Wie viele branches wird es wohl geben usw. Trotzdem wird wohl die Datenmenge gewaltig bleiben. Ganz davon abgesehen, das ein neues System angenommen werden muss. (Allein daran werden viele neue Ideen scheitern.)
Zentralisation ist eine Einschränkung
Wenn PubMed kraft der Dynamik seiner Bedeutung einst so viele Artikel zu verwalten gezwungen sein wird wie es Bücher in der Bibliothek von Babel zu geben scheint, wie sieht dann der Suchalgorithmus aus, der User M hilft, “sein” Dokument zu finden, insbesondere, falls es ihn befiele, aus Faulheit die Option “order by relevance” zu wählen?
Als ich noch bei http://www.frieling.de/
gearbeitet habe, habe ich eine solche Datenbank auch mal vorgeschlagen, aber damals war keienr davon so angetan.