Genomübergreifende Kooperationen
BLOG: Bierologie

Weil man es ja eigentlich gar nicht oft genug betonen kann: Wir sind alle transgene Organismen, denn unser Genom ist eigentlich nicht nur unseres. Das ist zumindest das Fazit, welches man aus der Endosymbiontentheorie ziehen kann. Seit dem Ende des 19. Jahrhunderts gibt es diese Theorie, doch wirklich groß geworden ist sie erst gegen Ende der 1960er Jahre, zusammen mit ihrer Fürsprecherin Lynn Margulis (die leider Ende des letzten Jahres verstorben ist). Im Endeffekt geht es darum das manche der Organellen – die man so in Zellen finden kann – ursprünglich mal freilebende Bakterien waren, sich allerdings im Laufe der Evolution irgendwann einen Platz in Zellen gesucht haben und es dort ganz kuschelig gefunden haben (und deshalb nicht wieder von dort verschwunden sind). Dafür helfen sie jetzt aber ihren Gastgebern zumindest mit ihrer Arbeitskraft. Eines der bekanntesten Beispiele für eine solche endosymbiontische Organelle dürften die Chloroplasten sein, welche nicht nur die Blätter grün werden lassen, sondern auch für die Photosynthese zuständig sind. Ein anderes Beispiel, was eben auch uns zu transgenen Organismen macht, sind die Mitochondrien. Diese Organellen, von denen die meisten irgendwann schon mal im Biologieunterricht als den “Kraftwerken der Zelle” gehört haben dürften, haben ihren Beinamen absolut verdient. Sie verbrauchen einen Großteil des Sauerstoffs den wir veratmen und produzieren über einen Stoffwechselweg, die oxidative Phosphorylierung, gute 95% der chemischen Energie die wir jeden Tag verbrauchen. Und für diesen relativ komplexen Stoffwechselweg benötigt man einen ganzen Haufen von Proteinen bzw. Protein-Untereinheiten.

Und in dem Zusammenhang können wir uns jetzt auch eines der starken Argumente für die Endosymbiontentheorie anschauen: Die Mitochondrien haben ein eigenes Genom, welches einen Teil der Gene enthält die man für diesen Stoffwechselweg braucht (und nur über die Eizellen der Mutter vererbt wird … So wie das Y-Chromosom also eine komplette väterliche Abstammungskette nachbildet, so kann man das für die mütterliche Seite mit Mitochondrien machen). Nach der Endosymbiontentheorie ist dieses mitochondriale Genom der Rest des ursprünglichen Bakteriengenoms. Allerdings hat dies mittlerweile nicht mehr alle Gene, die man für die Umwandlung von Sauerstoff zu nutzbarer Energie benötigt, denn irgendwo auf dem Weg haben sich viele der dafür nötigen Gene aus dem Mitochondrium verabschiedet. Allerdings haben sie uns nicht ganz verlassen, sondern sind in unser Kerngenom eingewandert, wo sie weiterhin eng mit den Mitochondrien zusammenarbeiten um für uns Energie zu produzieren. Diese enge Kooperationen der nukleären, also im Zellkern codierten, und der mitochondrialen Gene sollte deshalb zu einer Koevolution der dafür nötigen Gene führen: Wenn die Gene auf den beiden Chromosomen nicht zueinander passen, dann leidet die Fitness des Organismus der diese inkompatiblen Varianten trägt.
Eine Studie die genau diese Koevolution untersuchen soll wurde letztes Jahr von Patrick Flight und Kollegen veröffentlicht. Allerdings haben sie sich nicht den Menschen als Testobjekt angeschaut sondern einen Fisch aus den eierlegenden Zahnkarpfen, genauer gesagt die Art Fundulus heteroclitus. Dieser kommt unter anderem an der US-Ostküste an der Mündung des Hudson Rivers vor. Innerhalb dieser Art unterscheidet man noch zwei Unterarten, welche nördlich bzw. südlich der Flussmündung vorkommen und sich sowohl äußerlich unterscheiden, als auch von den physiologischen Eigenschaften die sie so verarbeiten können. Und auch genetisch unterscheiden sich die beiden Unterarten. Vor der Küste gibt es dann eine Hybridzone in der die beiden Unterarten sich miteinander verpaaren. Was dabei jedoch besonders spannend ist: Wenn man sich auf einer Karte anschaut wie die Verteilung der nukleären und mitochondrialen Genotypen ist, dann findet man das es keine einheitliche Zone für die Vermischung gibt, sondern das die Zonen ein wenig gegeneinander verschoben ist.
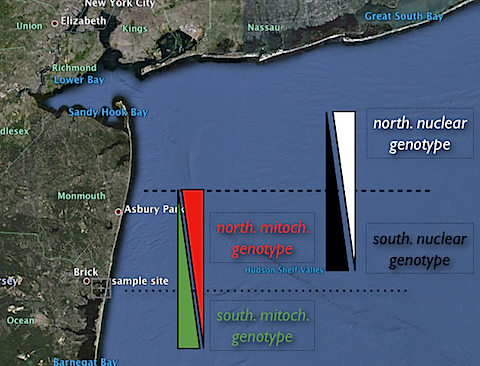
Auf der Karte sieht man die Verteilung der Genotypen. In Weiss und Schwarz die Verteilung der Genotypen für das Kerngenom. Je weiter man nach Norden geht desto mehr des nördlichen Genotyps findet man und desto weniger des südlichen Genotyps und umgekehrt. Genauso geht es mit Rot/Grün für den mitochondrialen Genotypen. Diese Verschiebung der beiden Zonen führt dazu das es einen Bereich gibt in dem man fast ausschliesslich den südlichen Genotyp für das Kerngenom, jedoch fast gleich verteilt den nördlichen und den südlichen mitochondrialen Genotyp findet. Das sorgt dafür, dass man zumindest in der Theorie ein ideales Feld hat um die Koevolution der beiden Genome zu untersuchen. Und entsprechend hat das Forscherteam an genau dieser Stelle seine Tiere für die Experimente gesammelt (In der Nähe von Brick auf der Karte oben). Für die anschliessenden Versuche hatten die Forscher 2 Thesen aufgestellt: 1. Der Genotyp des Mitochondriums sollte beeinflussen wie gut die Tiere Sauerstoffmangel-Stress überstehen können. 2. Der Genotyp des Mitochondriums sollte auch beeinflussen wie sich solcher Stress auf die Regulation der Gene auswirkt.
Um dies zu testen haben sie 2 Experimente durchgeführt: Als erstes haben sie die Tiere in 2 Gruppen eingeteilt und die eine Gruppe unter normalen Sauerstoffbedingungen und die andere unter Sauerstoffmangel (1/10 der Normalbedingung) gehalten. Dabei haben sie die Tiere über 25 Stunden beobachtet und dabei geschaut wie viele Tiere innerhalb der Beobachtungsperiode sterben. Anschliessend hat man sich die Genotypen der Tiere angeschaut und statistisch verglichen ob es Unterschiede im Überleben zwischen verschiedenen Gruppen gibt. Spannend dabei: Es macht für das Überleben keinen Unterschied welchen mitochondrialen Genotyp die Tiere haben. Genauso egal ist die Größe der Tiere. Alleine das Geschlecht hat einen signifikanten Einfluss auf die Überlebenswahrscheinlichkeit, die Frauen überleben länger.
Für den zweiten Versuch haben sie ihre Versuchsfische wieder in 2 Gruppen eingeteilt und die eine bei Normalsauerstoff gehalten, während die andere Gruppe in den gleichen sauerstoffarmen Bedingungen gehalten wurde. Nach 5 Stunden unter den Testbedingungen haben sie die Tiere getötet und RNA aus dem Lebergewebe isoliert. Mithilfe eines Mikroarrays hat sich das Team dann die Genregulierung für gut 6800 Gene angeschaut. Anschliessend hat man sich die Unterschiede in der Genregulierung für verschiedene Gruppen angeschaut und dabei neben dem mitochondrialen Genotypen auch den Einfluss des Geschlechts und auch dein Einfluss der Sauerstoffkonzentrationen verglichen. Zwischen den Geschlechtern findet man über 1000 Gene die unterschiedlich reguliert werden. Zwischen den Sauerstoffbedingungen finden sich immerhin noch über 600 Genem die sich in der Regulation unterscheiden. Darunter auch viele Gene die mit der sauerstoffverarbeitenden Funktion der Mitochondrien zu tun haben. Das ist natürlich nicht wirklich überraschend, immerhin unterscheiden sich die beiden Gruppen ja eben nur durch die vorhandene Sauerstoffmenge und dies sollte das Hauptziel von Genregulation sein. Nur zwischen den Genotypen wird werden die Unterschiede dann wieder schwach: Hier findet man nur noch 8 Gene die sich in der Regulation zwischen Nord und Süd unterscheiden und von diesen sind 5 vermutlich nur ein Artefakt des Experimentenaufbaus.
Es scheint in der Zusammenfassung also keinen so großen Einfluss auf das Überleben der Tiere und die Genregulation zu haben ob mitochondriale Gene und nukleäre Gene zusammenpassen. Allerdings muss man hier mit den Aussagen aufpassen: Als erstes ist die Genotypisierung der Tiere in dieser Publikation mit Vorsicht zu geniessen, da sie die nukleären Gene gar nicht typisiert haben um zu testen ob wirklich alle Tiere den “passenden” südlichen Genotyp haben. Und auch die Typisierung der Mitochondrien-Genome wurde nur über ein einfaches Fingerprinting gemacht. Von daher kann man nicht ausschliessen, dass man vielleicht die spannendere genetische Variation gar nicht mit erwischt hat bzw. diese nicht gezielt feststellen konnte. Damit kann es gut sein, dass die real vorhandenen Unterschiede verwischen. Außerdem merken die Autoren selbst auch an, dass es gut sein kann, dass die Unterarten sich noch nicht so weit genetisch ausdifferenziert haben als das man einen Unterschied feststellen könnte und es könnte genauso gut auch sein, dass man bei anderen Stressfaktoren für die Tiere (Salzgehalt im Wasser, Wassertemperatur,&) unterschiede zwischen den Genotypen finden könnte. Worüber ich mir auch noch Gedanken mache: In den Genregulations-Ergebnissen findet man auch, dass vor allem eine Sache bei Sauerstoffmangel herunter reguliert wird, nämlich die Produktion von Mitochondrien und auch die Expression der mitochondrialen Gene an sich. Und das könnte in meinen Augen auch dazu beitragen, dass man keine Unterschiede mehr finden kann: Wenn die Mitochondrien als Hauptreaktion auf Sauerstoffmangel einfach “ausgeschaltet” werden, dann dürfte es vermutlich auch relativ egal sein wie der Genotyp der Mitochondrien aussieht da sie einfach nicht benutzt werden. Ich bin gespannt ob andere Studien mit verbessertem Versuchsaufbau und anderen Methoden bei der Analyse von genomischer Koevolution helfen können.
FLIGHT, P., NACCI, D., CHAMPLIN, D., WHITEHEAD, A., & RAND, D. (2011). The effects of mitochondrial genotype on hypoxic survival and gene expression in a hybrid population of the killifish, Fundulus heteroclitus Molecular Ecology, 20 (21), 4503-4520 DOI: 10.1111/j.1365-294X.2011.05290.x
Foto Mitochondrium: Sterilgutassistentin, GNU General Public License


Mitochondrien
Schicker Aufsatz!
Als alter Anatom möchet ich aber darauf hinweisen, dass das (unser)gewöhnliches Bild vom Mitochondrium, so wie es in der ersten Abb. zu sehen ist, falsch ist. Das hab’ ich letztes Jahr von einem unserer Forscher hier gelernt – bei Interesse grabe ich die references aus.
Mitochondrium heisst verbatim “Fadenknötchen” – und so sehen sie auch aus. Die Dinger liegen (meist) nicht isoliert vor, sondern sind über dünne membranöse Brücken (wenn ich mich recht entsinne: Aussen- und Innenmembran) untereinander zu einem “Netz mit Knoten) (Knoten da, wo die Cristae/Tubuli sind) verbunden.
bei Interesse grabe ich…
Ja, bitte, unbedingt, die Sache interessiert mich sehr…
Gracias!
Referenzen auch finden!
Das interessiert mich ebenso wie der Artikel …
Fadenknötchen
Danke für die Korrektur, die References würden mich wirklich interessieren. Liegen diese Anordnung einfach an der ein wenig unvollständigen Biogenese nach Bakterienart? Also kommen durch eine nicht ganz vollständige Zellteilung zustande?
@ Bastian @ alle
War ja keine Korrektur, war nur eine Anmerkung. Was ich anmerkte, hab’ ich von Prof. Reichert (Excellence Cluster Macromolecular Complexes) hier an der Uni Ffm gelernt, bei seiner Antrittsvorlesung letztes oder vorletztes Jahr. Ich selbst verstehe wenig von der Sache.
Ich versuch’ morgen mal, ihn (Reichert) anzuhauen, und bitte ihn um ein Link auf
einen Review, das stell’ ich dann hierher. Hier ist schonmal seine HP, vielleicht wird man auch schon da fündig:
http://www.fmls-institute.de/index.php?id=60
Danke, in der Publikationsliste sind schon ein paar Titel dabei die so klingen als könnten die das richtige sein. Da schaue ich mich morgen mal in Ruhe um.
Was die Vermischung der Genome angeht, geht es ja noch irrer. Dass wir genetische Mosaike sind, ist ja leider noch nicht ganz in der Bevölkerung durchgesickert.
Jedenfalls musste ich als Ex-Pflanzenphysiologe sofort an die zwei plastidären RNA-Polymerasetypen denken: PEP, die Plastid-encoded Polymerase ist vom prokaryotischen Typus, deren Core-Struktur besteht aus vier Untereinheiten. NEP ist nukleär kodiert, monomer und ist wohl viralen (!!) Ursprungs. Diese muss nach Translation im Cytosol über entsprechende Trnasportmechanismen in den Plastiden importiert werden.
Das Irre ist, dass es auf dem Plastidengenom Gene gibt, die exklusiv von der nukleär codierten NEP transkribiert werden. Selbsteredend ist die Promotorstruktur bei Prokaryoten und Eukaryoten komplett unterschiedlich.