Die Evolution des genetischen Codes
BLOG: Bierologie


 Bastian sagt:
Bastian sagt:
Seit der Entdeckung der DNA durch Watson & Crick hat sich einiges getan. Mittlerweile ist das Prinzip wie aus dem Genom, dem Bauplan, nachher funktionierende Proteine entstehen bekannt. Doch wie genau es zum genetischen Code gekommen ist bleibt weiterhin offen. Aber genau dazu gibt es 2 Paper die sich mit diesem Thema beschäftigen.
Allerdings bin ich mir nicht ganz sicher wie viel Ahnung ihr von dem Grundmechanismus des genetischen Codes habt. Deshalb nochmal keine kurze Einführung:
Die Informationen in unserer DNA werden durch die 4 Basen Adenin, Thymin, Guanin, Cytosin und eben durch deren Abfolge hinterlegt. Auf dem DNA-Doppelstrang liegen jeweils Adenin & Thymin und Guanin & Cytosin sich gegenüber. Doch für jeden Bau eines Proteins seinen einzigen Bauplan, also sein Genom, aus der Hand zu geben wäre ganz schön unpraktisch und gefährlich.
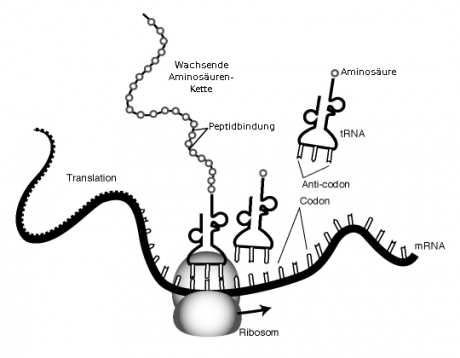
Deshalb wird im Schritt der Transkription die so genannte m(essenger)-RNA gebildet. Diese ist ein Negativ des kopierten Strangs, allerdings werden die T(hymin)-Basen in der mRNA durch U(racil) ersetzt. Wieso das so ist muss uns hier gerade gar nicht interessieren. Damit wäre der Schritt der Translation schon passiert. Mit dieser Kopie kann man nun anfangen sein Protein zu bauen. Und dieser Prozess nennt sich Transkription.
Jeweils 3 dieser Basen bilden ein sogenanntes Codon auch auch Triplett. In den Ribosomen paaren sich Codons der mRNA und die Komplementäre der so genannten tRNA an der wiederum die Aminosäuren hängen. Und diese Abfolge von Aminosäuren bildet das Protein. So weit zum Grundkurs Biologie.
Diese Codons aus jeweils 3 Basen bilden den genetischen Code. Durch die 4 verschiedenen Basen in Dreiergruppierungen ergeben sich 64 Kombinationen. Doch wie entstand dieser Code? Wie kommt es dass die Abfolge AUA für die Aminosäure Isoleucin codiert? Und warum sind die Codons 3 Basen lang und nicht länger oder kürzer? Und wieso überhaupt genau 4 verschiedene Basen und nicht mehr oder weniger?
Wieso die Codons eine Länge von 3 Basen haben wird dabei in dem ersten Paper was ich hier vorstellen möchte erörtert. Und zwar mit Hilfe einer Simulation. Die Theorie der Forscher war es dass die jetzigen Codons aus viel größeren Vorgängercodons evolviert sind. Denn in der jetzigen Form lässt sich die Transkription nicht ohne Ribosom erklären. Doch damit steht man vor einem typischen Henne & Ei-Problem.
Auf jeden Fall haben die Forscher sich ein nettes kleines Simulationsprogramm erdacht in dem sie mit größeren Codons starten und durch Selektion und Mutation schauen wo hin die Reise geht. Mit den genauen mathematischen Modellen will ich hier auch gar nicht wieder die breite Leserschaft langweilen, das Paper ist im Open Access-Journal PLoS ONE erschienen. Wer möchte kann dort direkt nachlesen.
Aber es zeigte sich dass die Codons im Laufe der simulierten Evolution immer kürzer werden bis sie sich irgendwo bei einer Länge von 3-4 einpendeln. Die Theorie der Forscher dazu ist folgende: Zu Beginn, als es den komplexen Apparat namens Ribosom zur Transkription noch nicht gab war es sinnvoll seine Information möglichst redundant zu codieren. So kann die eigentliche Information auch unter großem Rauschen noch herausgefiltert werden. Mit steigender Genauigkeit im Transkriptionsapparat wird es aber überflüssig die Informationen so sicher zu verpacken.
Im Gegenteil: Die Energie die man in das sichere Verpacken der Information setzt kann man jetzt viel besser in die eigentliche Reproduktion packen. Eine sehr hübsche Theorie dazu wie wir bei dem jetzigen Status bezüglich der Länge angelangt sein könnten. Doch das klärt noch nicht wieso der Code jetzt genauso aufgeteilt ist mit den Entsprechungen von Codon zu Aminosäure wie wir es heute haben.
Darüber hat Paul Higgs in Biology Direct vor einiger Zeit ein Paper veröffentlicht. Die codierenden Aminosäuren sind den Codons nämlich so zugeordnet dass die benachbarte Codons für Säuren mit ähnlichen physikalischen Eigenschaften codieren. Das hat den Vorteil dass bei Fehlern in der Translation die Fehler möglichst gering gehalten werden. Besser gesagt die Zuordnung zwischen Codons und Aminosäuren ist nicht rein zufällig. Irgendwie muss also in der Evolution der Code auf diese Formen hin optimiert worden sein.
Eine Theorie dazu bietet die Co-Evolution an. Während der Code zu Beginn nur wenige, verfügbare, Aminosäuren enthielt, kamen mit der Zeit mehr Aminosäuren dazu die dann nach und nach in den Code eingebaut wurden. Um diese Theorie näher zu beleuchten gab es dann wieder fleissige Modelle und mathematische Funktionen.
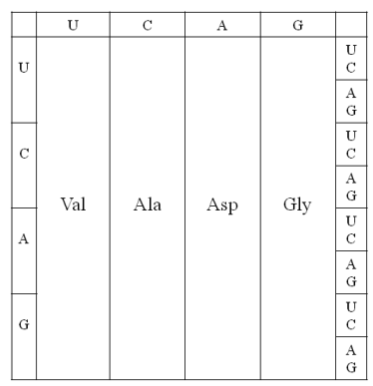
Die Theorie von Higgs geht davon aus dass die 4 abgebildeten Aminosäuren die ersten waren die eingebaut wurden. Und zwar so dass nur die zweite Stelle des Tripletts bestimmt welche Aminosäure codiert wird. Im Laufe der Evolution wurden dann die neuen Aminosäuren so eingebaut dass die Neuankömmlinge Positionen besetzen die vorher von möglichst physikalisch ähnlich gebauten Aminosäuren besetzt waren.
Dies bringt den großen Vorteil dass die bereits codierten Proteine durch diese Maßnahmen nicht völlig zerstört werden.
Dadurch sieht man auch dass die treibende Kraft für den Aufbau der Codes nicht die Verhinderung von Übersetzungsfehlern ist. Sondern die Selektion dahin geht mehr Aminosäuren zu codieren und damit auch mehr auch mehr Proteine bauen zu können.
Das sind alles schon sehr spannende Ergebnisse. Was mir immer noch ein wenig fehlt ist leider der Zusammenhang zwischen den 4 Basen die im genetischen Code vorkommen. Wieso genau 4? Wieso nicht mehr oder weniger? Wer dazu noch Ergebnisse kennt darf sich gerne in den Kommentaren austoben.
Higgs, P. (2009). A four-column theory for the origin of the genetic code: tracing the evolutionary pathways that gave rise to an optimized code Biology Direct, 4 (1) DOI: 10.1186/1745-6150-4-16
Baranov, P., Venin, M., & Provan, G. (2009). Codon Size Reduction as the Origin of the Triplet Genetic Code PLoS ONE, 4 (5) DOI: 10.1371/journal.pone.0005708


Die Antwort ist ganz einfach. Weniger als 4 Basen bedeutet 2 Basen, da es um die Ausbildung komplementärer Basenpaare geht. Mit 2 Basen würde man bei 20 Aminosäuren Basenquintetts statt Basentripletts benötigen (also 5 Basen pro Codon statt nur 3 Basen). Dann ist fraglich, wie man das stereochemisch hinbekommt, dass das Analogon zu einer tRNA mit 5 Basen an eine mRNA andocken kann. Die Anticodonschleife ist jedenfalls zu spitz dafür. Und generell sind RNA-Schleifen so verdreht, dass sie 5 Basen nicht so sortieren, dass sie sich an einen linearen Komplementärstrang anlagern könnten.
Das Analogon zu einem Ribosom müsste ebenfalls einen größeren Raum umgreifen, um zwei tRNA’s mit jeweils fünf Codon-Basen Platz bieten zu können. Auch das erfordert einen gewissen Mehraufwand an molekularem Baumaterial.
Mehr als 4 Basen würde wegen des Prinzips der komplementären Basenpaarung also 6 Basen bedeuten. Hier entstehen zwei wesentliche Schwierigkeiten: Entweder sind für 20 Aminosäuren Codon-Dupletts im Gebrauch – dann ergibt sich eine verringerte Fehlertoleranz, weil nur maximal 35 Dupletts als Repräsentanz von Aminosäuren zur Verfügung stehen (plus ein weiteres Duplett als Stopp-Codon).
Oder aber es sind Codon-Triplets im Gebrauch (was stereochemisch wiederum einfacher passen würde als Dupletts, da diese wiederum eine zu spitze Ausformung der Anticodonschleife erfordern) – dann müssten insgesamt 216 Tripletts belegt werden. Nehmen wir großzügigerweise an, dass sechs Tripletts als Stopp-Codon verwertet werden, bleiben immer noch 210 Tripletts, die mit Aminosäuren belegt werden müssen.
Bei 20 Aminosäuren würden pro Aminosäure im Durchschnitt etwa 10 Tripletts zur Repräsentation verwertet werden. (Im gebräuchlichen genetischen Code sind es nur etwa 3!) Bei einer so hohen Fehlertoleranz würde sich eine zu geringe Zahl an Mutationen ergeben, die sich phänotypisch auswirken könnte, so dass die Variationsbreite der Organismen auf einem sehr schmalen Bereich begrenzt bliebe. Dadurch erhöht sich aber die Anfälligkeit gegenüber selektiven Drücken, so dass statt Evolution das Aussterben die Folge ist.
Hinzu kommt der Mehraufwand an Stoffwechsel, um die benötigten 6 Basen für das Genom zur Verfügung zu stellen. Mit Xanthin und Diaminopyrimidin stünden zwar zwei weitere Basen als gangbare Kandidaten zur Verfügung, die (im Falle von Xanthin) auch als Intermediat im Stoffwechsel üblicherweise anfallen, aber diese müssten eben von vom Stoffwechsel abgezweigt werden, um sie als Genom-Basen zu verbauen.
Falls es also in der fernen Vergangenheit Ansätze zur Entstehung eines sechsbasigen Genoms gegeben haben sollte, wurden diese durch die wesentlich fitteren Varianten mit vier Basen verdrängt. Ebenso trifft das auf eventuelle zweibasige Genom-Anläufe zu.